在众多新能源项目中,生物质能源因兼顾废物处理与能源回收而获得重视[1].许多农牧业有机废物,例如秸秆[2]、家畜粪便[3]等,均已被证实能够通过中温或者高温厌氧消化的方式转变为生物质气体.但是,直接以上述有机废物作为底物进行厌氧消化的效率往往较低[4],限制了该技术的推广.近年来,发现对有机废物进行适当的预处理可以大幅提高后续厌氧消化的效率[5].例如,本团队曾借助“热-酶联合预水解法”成功实现了对啤酒糟和猪粪的预处理,相比直接处理上述底物,该预处理方法将厌氧消化的效能提高了5~7倍[6].
对于应用规模的厌氧消化反应器,在实际运行过程中往往会接纳不同类型的有机物.有研究指出,底物类型的转变对厌氧消化过程的影响较大,主要体现在对厌氧消化产甲烷效率[7]以及对微生物种群组成的的影响[8-9].目前关于底物类型对厌氧消化过程的效能及微生物种群影响方面的报道较少.本研究以经过“热-酶联合预水解”后的啤酒糟(brewery spent grain hydrolysates,BSGH)和猪粪(pig manure hydrolysates,PMH)为处理对象,以升流式厌氧膨胀床(expanded granular sludge bed,EGSB)作为厌氧消化过程的载体,考察依次以BSGH和PMH作为有机底物时EGSB反应器的厌氧消化效能,包括化学需氧量(chemical oxygen demand,COD)的去除情况、挥发性有机酸(volatile fatty acids,VFAs)的累积以及产甲烷的效率.此外,借助高通量测序以及定量PCR等分子生物学手段考察了反应器内的微生物群落对底物变化的响应.
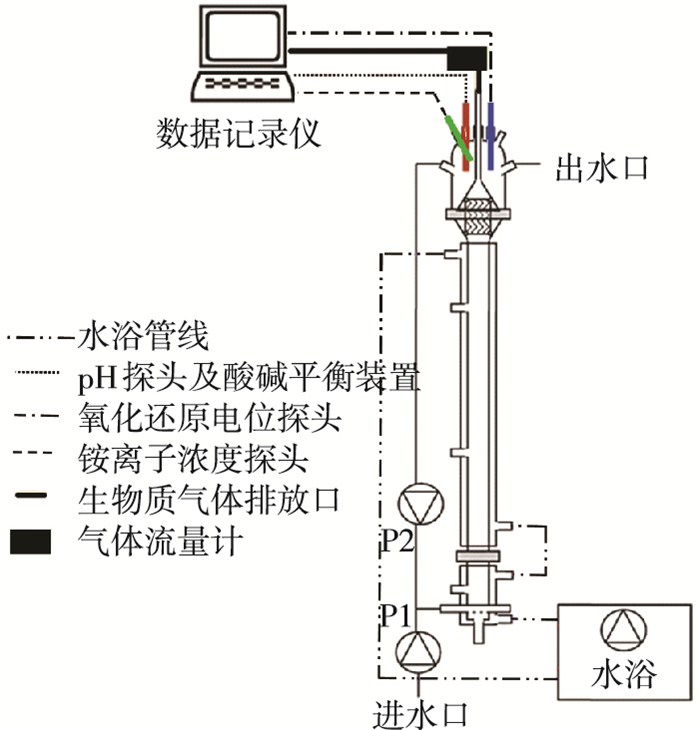
1 实验 1.1 EGSB反应器EGSB反应器主体由双层玻璃制成(见图 1),有效容积为3.8 L.利用水浴维持反应器内的温度(35±1) ℃.蠕动泵P1为进水泵,P2为出水回流泵.通过P2高速运转实现反应器内的上升流速8 m/h,从而保障反应器内液体的膨胀状态.借助置于反应器顶部的pH、ORP和铵离子质量浓度探头实时监测上述3个指标的数值变化并通过数据记录仪存储数据.为了防止极端pH造成反应器的不稳定,通过酸碱平衡装置将pH维持在6.9~7.1.具体做法是当反应器内pH低于6.9时,利用微量蠕动泵向反应器内滴加0.1 mmol/L的NaOH溶液,当反应器内pH高于7.1时,滴加0.01 mmol/L的HCl溶液.采用德国Ritter公司生产的MGC-1 PMMA型气体流量计对生物质气体的产生量进行实时监测,并借助安捷伦公司生产的HP7890A型气相色谱检测生物质气体中甲烷的质量分数,计算产甲烷速率.采用某土豆深加工厂污水厌氧处理设备的成熟颗粒污泥作为接种污泥.
|
图 1 EGSB反应器装置 |
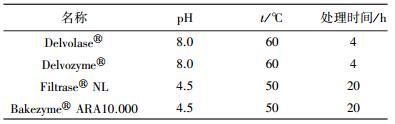
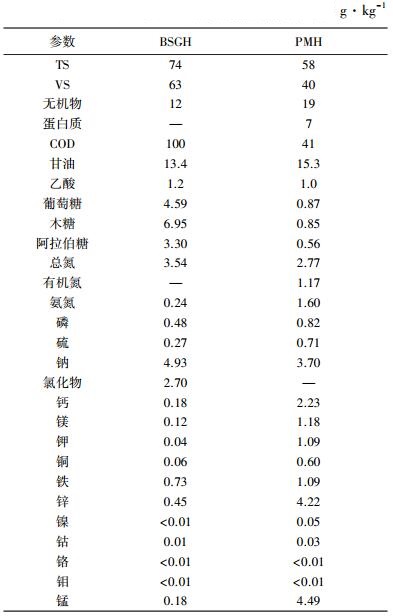
采用热-酶联合预水解方法对啤酒糟和猪粪分别进行预处理,步骤见文献[6].对啤酒糟的预处理过程主要包含以下步骤:首先,对啤酒糟在pH 10.7、90 ℃条件下热处理4 h;依次采用荷兰DSM公司生产的Delvolase®蛋白酶(单独使用)、Filtrase® NL纤维素水解酶和Bakezyme® ARA10.000半纤维素水解酶(同时使用)对底物进行酶解,具体条件如表 1所示;最后,为了排除无机颗粒或难降解残渣对后续厌氧消化的不利影响,采用瑞士Sefar公司生产的SEFAR TETEX® 05-6-456K型聚丙烯网纱对水解后的混合液进行过滤并回收滤出液(即BSGH).对猪粪的预处理过程与啤酒糟的处理过程相似,不同点在于将Delvolase®蛋白酶与Delvozyme®蛋白酶同时使用.BSGH和PMH底物的主要成分如表 2所示.
| 表 1 酶水解过程的最优条件 |
| 表 2 经热-酶预处理后啤酒糟和猪粪的成分 |
采用德国MERCK公司生产的COD和氨氮质量浓度测定试剂盒及TR420/NOVA60型分光光度计对上述指标进行检测.溶解性COD样品测定前,采用英国Whatman公司生产的Spartan 30型0.45 μm滤膜对样品进行过滤.挥发酸和甲烷成分的测定分别由配备FID检测器和TCD检测器的安捷伦HP7890型气相色谱完成.上述指标的测定过程采用平行样品测定以减少仪器误差.总COD去除率、溶解性COD去除率及有机物甲烷化率的计算公式如下:

|
式中:COD进水、COD出水分别为进、出水总COD,g/L;溶解性COD进水、溶解性COD出水分别为进、出水溶解性COD,g/L;2.66为室温下单位体积甲烷所对应的化学需氧量系数;V甲烷为产甲烷体积,L/d;Q为反应器的进水流量,L/d.
1.4 分子生物学分析方法为了表征反应器污泥中细菌和古细菌的数量及其群落结构,对接种污泥以及第8、34、56和76天的污泥样品进行DNA提取和定量PCR、高通量测序分析.此外,为了考察处于反应器不同高度的污泥中细菌和古细菌的数量,在反应器运行非常稳定阶段(第56天)从反应器上、中、下3个取样口分别取样.
利用美国MoBio实验室生产的MoBioUltraClean细菌DNA提取试剂盒对污泥样品进行DNA提取,提取后的DNA样品一部分用于454-焦磷酸高通量测序,另一部分用于定量PCR分析.定量PCR实验采用美国ABI公司生产的ABI 7500型qPCR分析仪完成,采用的特异性引物如表 3所示.引物和SYBR Premix Ex Taq Kit预混装试剂盒购自工生物工程(上海)股份有限公司,依照预混装试剂盒使用说明书配置反应体系.细菌qPCR分析过程的条件为:95 ℃预变性60 s,95 ℃循环变性15 s,53 ℃退火复性30 s,72 ℃延伸45 s,进行40个循环;古细菌qPCR分析过程的条件为:95 ℃预变性60 s,95 ℃循环变性10 s,61 ℃退火复性30 s,72 ℃延伸45 s,进行40个循环.全部定量PCR待测样品均经过3次重复测定取平均值.另一部分DNA样品送美国R & T实验室进行454-焦磷酸测序(罗氏454 GS-FLX系统).选择Universal正向引物序列U515F (‘5-GTGYCAGCMGCCGCGGTA A-3’)和反向引物序列U1071R (‘5-GAR CTGRCGRCRRCCATG CA-3’)合成Barcode引物,该引物可覆盖90%以上的细菌和古细菌[10].测序结束后,借助微生物生态学集成软件QIIME(1.7.0版本软件)对原始文件进行后续生物信息学分析[11].
| 表 3 qPCR分析采用的特异性引物及其序列 |
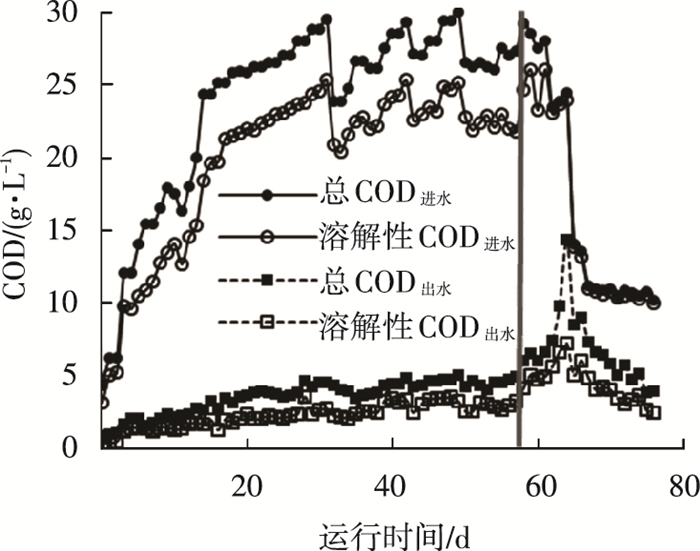
整个实验阶段共持续76 d,分为两个阶段.第一阶段采用BSGH底物运行57 d,第二阶段以PMH为底物继续运行19 d,图 2~6及图 9中标记出底物类型转变的日期界限.整体看,第一阶段的总COD去除率平均值为85%,溶解性COD去除率平均值为89%,第二阶段上述两个指标的平均值分别为55%和70%,均低于第一阶段.通过图 2可以看出,当进水有机底物从BSGH转变为PMH后,出水的总COD和溶解性COD均升高,即使进水总COD由25 g/L降至10 g/L后,出水COD仍高于第一阶段的水平.当改变有机底物后,出水总COD和溶解性COD均出现峰值,分别为14.3和7.2 g/L.
|
图 2 进出水COD变化 |
|
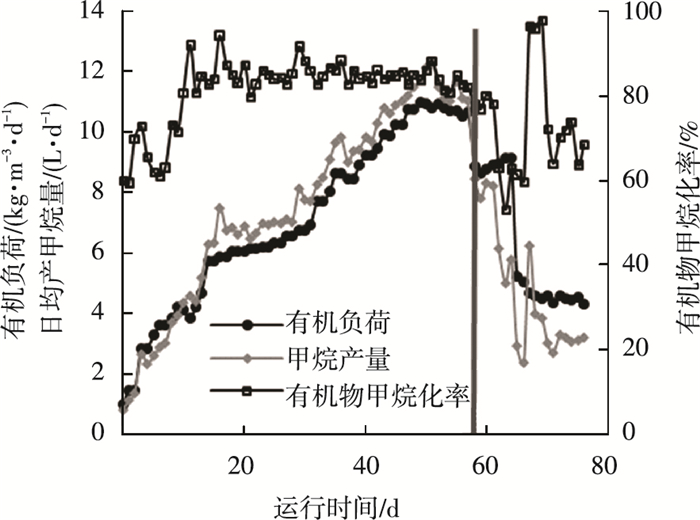
图 3 有机负荷、甲烷产量和有机物甲烷化率变化 |
|
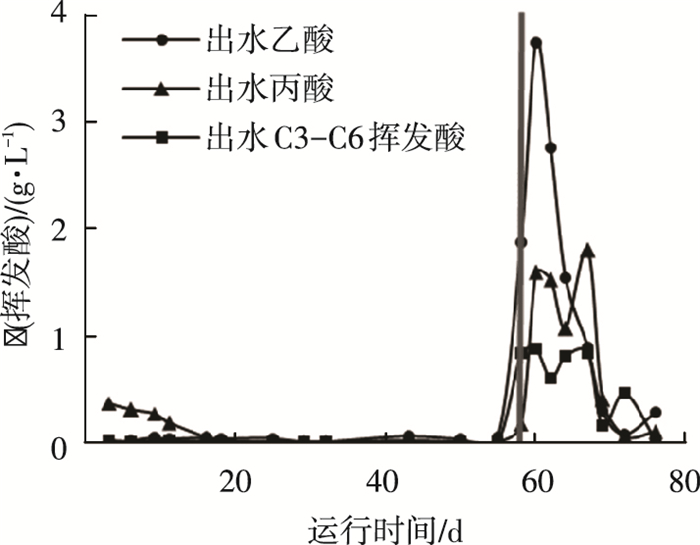
图 4 挥发性有机酸质量浓度变化 |

|
图 5 细菌和古细菌数量变化 |
|
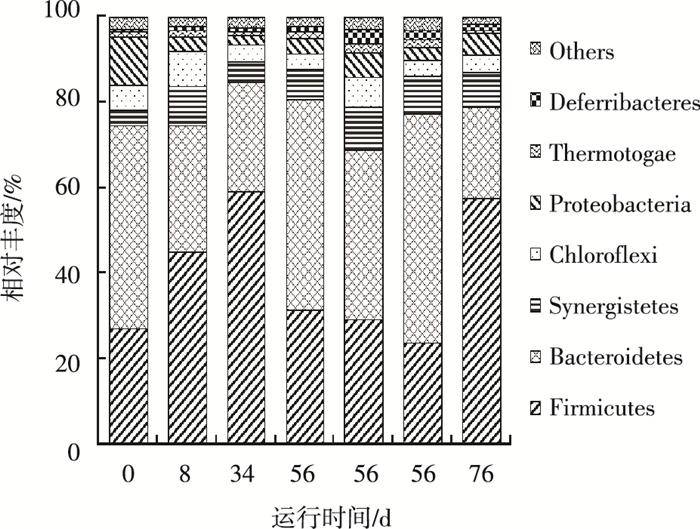
图 6 反应器中细菌菌门分布 |
|
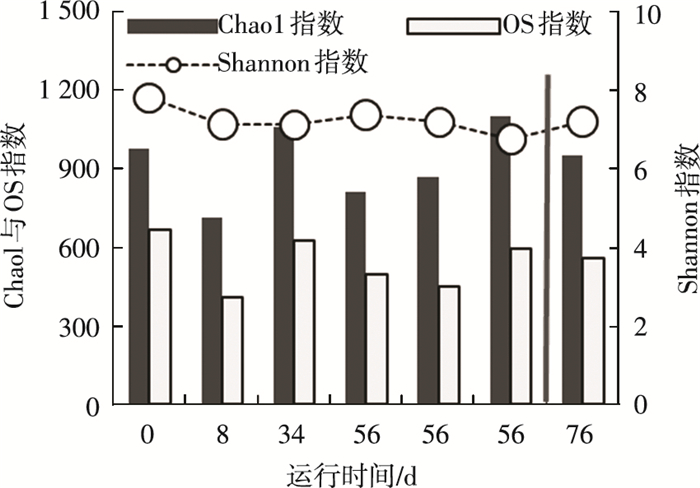
图 9 群落的α生物多样性指标变化 |
由图 3可以看出,在实验的第一阶段,施加BSGH底物的有机负荷由1.0 kg/(m3·d)逐渐提升至10.9 kg/(m3·d).随着有机负荷的提高,甲烷产量由0.8 L/d提高至12.1 L/d.在启动的最初10 d左右,有机物甲烷化率处于不稳定期,在60%~70%波动.但随着有机负荷逐步提升,反应器进入稳定运行阶段,有机物甲烷化率平均值达85%.这意味着在EGSB稳定运行阶段,BSGH中85%的有机物最终转变为甲烷,其余部分则转变为细胞物质、CO2或作为电子供体参与其他生化过程.进入第二阶段后,反应器运行效果变差,将有机负荷由10.9 kg/(m3·d)调低至8 kg/(m3·d),而后进一步降低至4 kg/(m3·d).在逐步降低有机负荷过程中,反应器的效能并无恢复的趋势,甲烷产量逐步降至3 L/d,有机物甲烷化率也降至60%左右.
由图 4可以看出,当转变有机底物类型时,反应器内出现明显的挥发性有机酸累积情况,特别是乙酸质量浓度从第一阶段的低于50 mg/L跃升至3 700 mg/L,同时丙酸和丁酸也出现不同程度的积累.大幅降低有机负荷水平以后,反应器内的挥发酸积累情况有所缓解.由于对反应器实施了pH监控及平衡措施,反应器的pH始终保持在6.9~7.0.值得注意的是,在第二阶段挥发酸积累的同时,大量的颗粒污泥开始解体并随出水流失.在第一阶段反应器内的TSS和VSS质量浓度为20.0和21.5 g/L,而第二阶段TSS和VSS质量浓度下降到8.8和7.4 g/L.不难发现,挥发酸的积累和污泥大量流失的现象伴随着甲烷产量及有机物甲烷化率骤减现象同期出现,表明厌氧污泥的性质和活性受底物类型转变的影响.微生物数量和群落结构的变化可能是导致这一现象的内因.
2.2 底物类型对微生物数量的影响反应器启动1周后,污泥中细菌和古细菌的数量相比于接种污泥(第0天样品)均有降低(如图 5所示).随着系统逐步稳定,细菌和古细菌数量逐步升高.第56天从反应器上、中、下3个取样口分别获得的样品(图 5中第56天从左至右依次代表上、中、下取样口样品)的分析结果表明,细菌和古细菌的数量从上至下逐渐升高,反应器底部的细菌和古细菌数量分别是顶部数量的7倍和10倍.当转变底物类型后,细菌的数量(图 5中第76天样品)保持稳定,但古细菌的数量减少61%.由于产甲烷菌占到了古细菌群落的95%以上,可以推测产甲烷菌数量因底物类型的转变而大幅降低,这也与同时期挥发酸的积累(图 4)和甲烷产量的大幅降低现象(图 3)相关.
由于施加BSGH和PMH底物的有机负荷相同,推测最可能造成产甲烷菌数量骤减的原因是PMH底物中的某些成分抑制了产甲烷菌的活性.对比两种底物的成分(如表 2所示),PMH中氨氮的质量浓度为1.6 g/L,而BSGH中氨氮质量浓度仅为0.24 g/L,在有机负荷相同的情况下,PMH中氨氮和总氮的质量浓度分别是BSGH的16倍和2倍.据文献报道,高氨氮往往容易对厌氧消化过程,特别是产甲烷过程产生抑制[12-13].
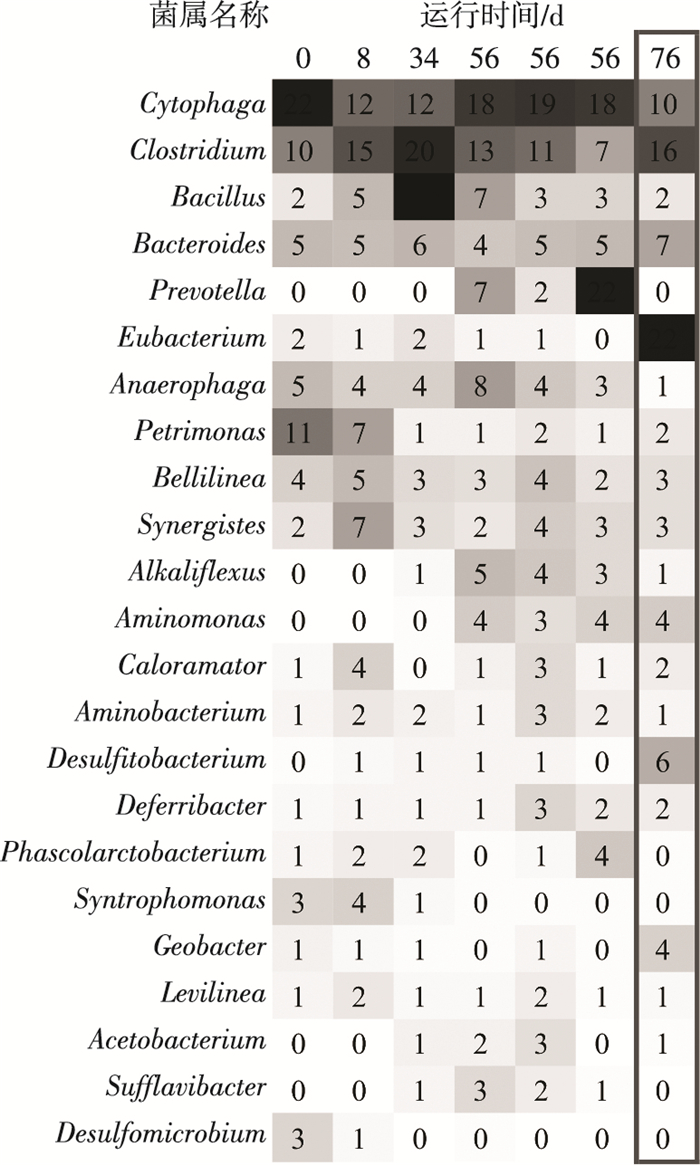
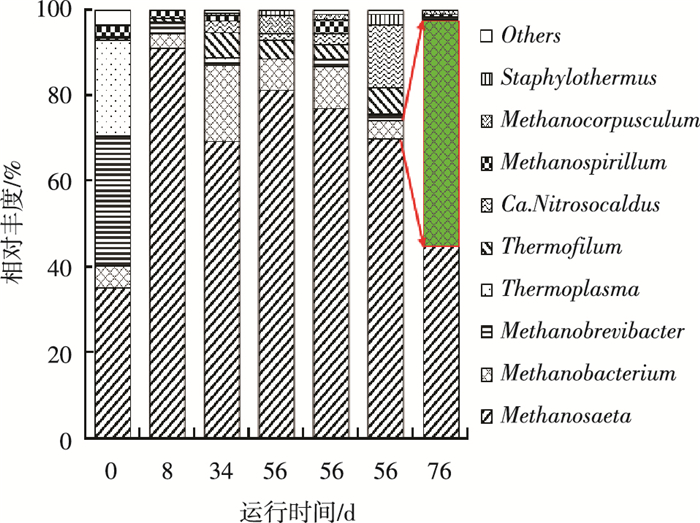
2.3 微生物群落结构的变化EGSB反应器中Firmicutes菌门和Bacteroidetes菌门是最占优势的两个细菌菌门(如图 6所示).在接种污泥中,Firmicutes和Bacteroidetes菌门的相对丰度分别为27%和47%,引入BSGH后,Firmicutes菌门的相对丰度逐渐升高至60%,而Bacteroidetes菌门的丰度则降至26%.至第56天时,二者的相对丰度分别为29%和39%.从不同高度位置的污泥中菌门分布情况看,3个样品的优势菌门分布较一致.底物类型的改变对细菌菌属分布同样产生较大影响.如图 7所示,在第一阶段占优势的细菌菌属为Cytophaga、Clostridium以及Bacillus,它们在该阶段的平均相对丰度之和达35%以上.而三者在第二阶段的丰度之和只有27%左右.Eubacterium成为第二阶段最占优势的细菌菌属,其相对丰度从第一阶段的平均1%跃升至第二阶段的22%.底物类型的改变同样对产甲烷菌群产生较大影响.如图 8所示,古细菌菌属的分布在第一阶段保持相对稳定,以Methanosaeta为最优势菌属,其相对丰度平均值达77%.转变有机底物类型后,Methanosaeta的相对丰度大幅减少至45%,同时Methanobacterium的相对丰度由8%提升至53%.
|
图 7 反应器中细菌菌属分布 |
|
图 8 反应器中古细菌菌属分布 |
Methanosaeta是严格的乙酸利用型产甲烷菌,而和Methanobacterium则是严格的氢气利用型产甲烷菌.Wilson等[14]认为,高氨氮质量浓度对乙酸利用型产甲烷菌的抑制作用强于对氢气利用型产甲烷菌的抑制作用.因此推测,当底物转变为氨氮质量浓度极高的PMH后,反应器内的乙酸利用型产甲烷菌Methanosaeta受到抑制,数量也大幅减少(如图 5所示),导致Methanobacterium的相对丰度提高(如图 8所示).由于乙酸利用型产甲烷菌Methanosaeta的大量流失,反应器内产甲烷的途径发生改变,导致细菌菌群同样发生变化,例如,主要的产乙酸细菌Cytophaga的丰度降低.由于作为厌氧过程重要中间代谢产物的乙酸分子无法及时被利用,其他产酸途径同样受到抑制,导致丙酸和丁酸等挥发酸的积累,因此,诸如Clostridium和Eubacterium这类对挥发酸更耐受的菌属逐渐占优势(如图 7所示).
2.4 种群的生物多样性变化底物类型的改变对微生物群落的生物多样性影响不明显.如图 9所示,表征α多样性中物种丰富度指标的Chao1和OS指数以及表征均匀度与丰富度综合指标的Shannon指数均比较稳定,特别是改变底物类型后上述3个指标未发生明显变化.过去的研究表明,厌氧消化系统内微生物群落的均匀度比较稳定[15],且一般微生物多样性指标的变化非常缓慢[16],因而, 本研究中反应器运行的时间尚不足以造成微生物多样性产生明显变化.
3 结论1) 有机底物类型的变化对厌氧消化反应器的运行效能产生较大影响,COD去除率降低约40%,甲烷产量减少75%,有机物甲烷化率降低25%,且反应器内出现较严重的挥发酸积累情况,出水中乙酸质量浓度由50 mg/L跃升至3 700 mg/L.
2) 底物类型的转变导致产甲烷菌数量大幅减少61%.由于乙酸利用型产甲烷菌Methanosaeta耐受高氨氮质量浓度的能力极弱,当底物更换为含有较高氨氮的PMH后,Methanosaeta的活性及生长受到抑制,导致产甲烷菌数量骤减,间接导致产酸菌群紊乱,最终造成反应器效能的降低.
3) 对于实际厌氧消化工艺而言,当不可避免变更有机底物类型时,宜降低底物的有机负荷,并严密监测系统内挥发酸的积累和甲烷产量等指标,以避免因新底物中的某些组分对微生物种群产生抑制而导致反应器效能的下降.
| [1] |
MAO C, FENG Y, WANG X, et al. Review on research achievements of biogas from anaerobic digestion[J]. Renewable and Sustainable Energy Reviews, 2015, 45: 540-555. DOI:10.1016/j.rser.2015.02.032 |
| [2] |
ZHANG J, GUO R, QIU Y, et al. Bioaugmentation with an acetate-type fermentation bacterium Acetobacteroides hydrogenigenes improves methane production from corn straw[J]. Bioresource Technology, 2015, 179: 306-313. DOI:10.1016/j.biortech.2014.12.022 |
| [3] |
ZHAI N, ZHANG T, YIN D, et al. Effect of initial pH on anaerobic co-digestion of kitchen waste and cow manure[J]. Waste Management, 2015, 38: 126-131. DOI:10.1016/j.wasman.2014.12.027 |
| [4] |
BIDART C, FROHLING M, SCHULTMANN F. Livestock manure and crop residue for energy generation:macroassessment at a national scale[J]. Renewable & Sustainable Energy Reviews, 2014, 38: 537-550. |
| [5] |
ZHANG C, SU H, BAEYENS J, et al. Reviewing the anaerobic digestion of food waste for biogas production[J]. Renewable & Sustainable Energy Reviews, 2014, 38: 383-392. |
| [6] |
WANG H, TAO Y, TEMUDO M, et al. An integrated approach for efficient biomethane production from solid biowastes in a compact system[J]. Biotechnology for Biofuels, 2015, 8: 62. DOI:10.1186/s13068-015-0237-8 |
| [7] |
李秀芬, 胡庆昊, 陈坚. 不同底物条件下金属离子螯合剂对厌氧消化的影响[J]. 环境科学, 2009, 30(6): 143-146. |
| [8] |
YENIGUN O, DEMIREL B. Ammonia inhibition in anaerobic digestion:a review[J]. Process Biochemistry, 2013, 48: 901-911. DOI:10.1016/j.procbio.2013.04.012 |
| [9] |
ZIGANSHIN A M, LIEBETRAU J, PROTER J, et al. Microbial community structure and dynamics during anaerobic digestion of various agricultural waste materials[J]. Applied Microbiology Biotechnology, 2013, 97(11): 5161-5174. DOI:10.1007/s00253-013-4867-0 |
| [10] |
WANG Y, QIAN P. Conservative fragments in bacterial 16S rRNA genes and primer design for 16S ribosomal DNA amplicons in metagenomic studies[J]. PLoS One, 2009, 4(10): e7401. DOI:10.1371/journal.pone.0007401 |
| [11] |
CAPORASO J G, KUCZYNSI J, STOMBAUGH J, et al. QⅡME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [12] |
YENIGUN O, DEMIREL B. Ammonia inhibition in anaerobic digestion:a review[J]. Process Biochemistry, 2013, 48(5/6): 901-911. |
| [13] |
WANG X J, LU X G, LI F, et al. Effects of temperature and carbon-nitrogen (C/N) ratio on the performance of anaerobic co-digestion of dairy manure, chicken manure and rice straw:focusing on ammonia inhibition[J]. PLoS One, 2014, 9(5): 15-20. |
| [14] |
WILSON C A, NOVAK J, TAKACS I, et al. The kinetics of process dependent ammonia inhibition of methanogenesis from acetic acid[J]. Water Research, 2012, 46(19): 6247-6256. DOI:10.1016/j.watres.2012.08.028 |
| [15] |
CARBALLA M, REGUEIRO M L, LEMA J M. Microbial management of anaerobic digestion:exploiting the microbiome-functionality nexus[J]. Current Opinion in Biotechnol, 2015, 33: 103-111. DOI:10.1016/j.copbio.2015.01.008 |
| [16] |
MADSEN M, HOLM-NIELSEN J B, ESBENSEN K H. Monitoring of anaerobic digestion processes:a review perspective[J]. Renewable and Sustainable Energy Reviews, 2011, 15: 3141-3155. DOI:10.1016/j.rser.2011.04.026 |
 2016, Vol. 48
2016, Vol. 48