2. 哈尔滨工业大学市政环境工程学院,哈尔滨 150090;
3. 东北电力大学 化学工程学院,吉林 吉林 132012
2. School of Municipal and Environmental Engineering, Harbin Institute of Technology, Harbin 150090, China;
3. College of Chemical Engineering, Northeast Electric Power University, Jilin 132012, Jilin, China
水质生物稳定性关系到饮用水水质安全.在给水管网系统中,不受控制的生物再生过程会导致水质恶化,卫生指标超标、口感不佳,还会导致过滤器堵塞、管道生物腐蚀等[1].出厂水水质若不稳定还会为致病菌增殖提供相对丰富的微环境,恶化管网水质,危害人体健康[2].因此,提高饮用水水质的生物稳定性、控制微生物再繁殖具有重要意义.
传统的饮用水水质生物稳定性评价方法主要集中于评价水体的微生物生长潜能.现行饮用水水质标准对生物稳定性方面提出更严格的要求,同时,新兴分析方法尤其是分子生物学方面分析技术的崛起促进了生物稳定性评价方法的发展.本文针对生物稳定性评价方法进行了综述与分析,包括生物稳定性评价指标及传统评价方法、基于流式细胞术改进的新兴评价方法、变性梯度凝胶电泳和焦磷酸测序等新兴分子生物学评价方法.从测定原理及应用实例等方面归纳总结了不同评价方法的优缺点以及今后的发展趋势.
1 生物稳定性评价指标及其传统评价方法目前,国内外用于评价饮用水水质生物稳定性的指标主要有基于培养法的异养菌平板计数(heterotrophic plate counts, HPC)、可生物降解有机碳(biodegradable dissolved organic carbon, BDOC)、可生物同化有机碳(assimilable organic carbon, AOC),不依赖培养的总细胞浓度(total cell concentration, TCC)和三磷酸腺苷计数(adenosine tri-phosphate, ATP)等.
1.1 异养菌平板计数异养菌平板计数(HPC)是指在特定条件下培养细菌,利用显微镜观察细菌数来评价水体的微生物生长潜能.HPC作为微生物参数来评估和监测饮用水水质已有100余年的历史,目前仍是国际上评价水质微生物量的标准方法[3-5].
传统的HPC测定采用倾倒接种法,以平板计数琼脂(plate count agar, PCA)为培养基,培养接种细菌并观察细菌生长数量.该方法技术稳定,但PCA具有选择性,不能检出产色素菌,而且倾倒法存在热冲击、转种培养不便等缺点[6].改进后的R2A培养基营养成分较PCA丰富且含量低,适合产色素菌生长,还能够促进受损细菌的恢复性再生长、提高饮用水中细菌总数检出率,已成功用于检测饮用水水样、管道内壁生物膜、污损反渗透膜内的异养菌数量[7].滤膜计数法以m-SPC为培养基,可大大提高接种量,特别适用于分析低浊度水样,但检测结果受滤膜质量等因素影响[8].
然而,越来越多的研究指出,HPC方法检测所得的微生物量只占实际微生物量的一小部分,难以准确反映水体中微生物量的变化[9-12].这是因为天然水环境中的大多数微生物不能用平板法进行培养,而且水体中还可能存在大量的自养菌.此外,HPC方法的分析结果受培养基的种类、培养时间、培养温度影响较大.但HPC方法操作简单,检测成本低,且已被广泛接受,因此,仍被广泛用于饮用水水质微生物风险评估[12].
1.2 可生物降解有机碳可生物降解有机碳(BDOC)是溶解性有机碳(dissolved organic carbon, DOC)的一部分,指水体中能被异养菌代谢和利用的溶解性有机碳,是20世纪末评估水质生物稳定性的主要指标之一[13].BDOC的测定方法由Servais等[14]发明,将待测水样经2 μm的滤膜过滤去除水样中微生物后接种一定量同源细菌,在恒温条件下培养并测定培养前后DOC的差值.
Servais等[15]认为当出厂水BDOC≤0.16 mg/L时,即使管网中没有消毒剂残余也会保证其水质生物稳定性.Volk等[16]认为水温分别在20和15 ℃时,对应的BDOC值不高于0.15和0.3 mg/L时,都能保证水质生物稳定性.van der Kooij等[17]却认为BDOC指标不能用于预测管网水的微生物再繁殖水平,因为BDOC参数与异养菌计数参数几乎没有相关性.Kaplan等[18]认为BDOC侧重于预测出厂水需氯量及消毒副产物的潜在生成量,可生物同化有机碳(AOC)更能够评价微生物的再生水平.
事实上,BDOC的检测方法限制了其作为生物稳定性评价指标的应用.BDOC的检出限由总有机碳分析仪决定,其测得的初始总有机碳(total organic carbon, TOC)值和最终TOC值非常相近,使得BDOC的检出限偏高(0.1~0.2 mg/L),远低于AOC的测定精度[19].另外,BDOC最终矿化成CO2或同化成生物碳,因此,只有部分BDOC用于微生物再繁殖.目前,BDOC主要用来预测和衡量水处理单元特别是生物处理单元对有机物的去除效率,以及预测出厂水需氯量和消毒副产物的潜在生成量.
1.3 可生物同化有机碳可生物同化有机碳(AOC)是指溶解性有机碳中能被微生物同化成自身菌体的部分,代表了最容易被生物降解的有机碳[20],是近20年来国内外评价饮用水水质生物稳定性的主要指标之一.van der Kooij等[17]发现出厂水AOC浓度与管网中的异养菌数有很大的相关性,最先提出以AOC作为水质生物稳定性评价指标,并得到一个指导性的结论:当AOC质量浓度低于10 μg/L乙酸碳时,异养细菌几乎不能生长,饮用水水质生物稳定性良好.LeChevallier等[21]认为在余氯质量浓度大于0.5 mg/L或者氯胺质量浓度大于1 mg/L的管网系统中,当AOC质量浓度低于50~100 μg/L时,大肠杆菌的生长受到限制.
最早的AOC测定方法由van der Kooij提出,即在巴氏灭菌后的待测水样中接种50~500 cfu/mL荧光假单胞菌P17,于15 ℃恒温培养期间通过平板计数获得培养的细菌数,再根据生长因子换算成乙酸碳浓度[22-23].之后,Kooij又增加一种可利用草酸的螺旋菌NOX作为测试菌种,将AOC的测定精度提高到微克级,但Kooij的测定方法操作复杂、实验周期长.LeChevallier等[24]将培养温度提高到25 ℃,接种浓度增加至104 cfu/mL,两种测试菌在培养2~3 d后达到稳定期,改进后的培养方法大大地缩短了培养时间,在AOC测定中被广泛采用.
但是,近年来有学者质疑AOC作为评价生物稳定性指标的客观性.研究表明,每消耗10 μg/L的有机碳,会导致每毫升水体中104~105个细菌生长[10, 25-26].即当AOC浓度低于检出限时,仍然可能有大量的微生物增长.且AOC只是被异养菌消耗,当水体中由于自养菌(如一些反硝化细菌和大多数氨氧化细菌)的繁殖而引起水质生物稳定性失衡时,AOC指标便失去指导意义[27].事实上,AOC依然是控制及优化某些特定水处理过程的有效评价参数,同时也是出厂水进入管网前的主要评价指标.Lautenschlager等[27]认为将AOC与总细胞浓度(TCC)、三磷酸腺苷(ATP)等评价指标结合,多参数评价水质生物稳定性,在保证水质安全和稳定方面更具指导意义.
1.4 其他综合评价指标总细胞浓度(TCC)指单位体积水体中存在的细菌数量,是表征微生物数量的直接参数,通过细菌直接计数测得.与表征可培养细菌总数的HPC相比,TCC还包括了“活的不可培养”的细菌数,因而能更为全面地评价水体中的微生物水平.早期TCC的测定主要借助显微镜来进行细胞观察计数,随后落射荧光显微术的日趋成熟推动了细菌直接计数技术的发展,但仍存在仪器操作、显微镜使用复杂,多种染色程序、数字图像分析耗时等缺点[28],限制了TCC作为生物稳定性评价指标的大规模应用.
三磷酸腺苷(ATP)是微生物细胞的“能量货币”,存在于所有活细胞中.研究表明,水中ATP含量与活细胞数量呈正相关关系,通过测定ATP含量可间接反映水环境中的活性生物量[29].ATP计数法即利用生物发光技术来测定微生物中的ATP量,测试原理是在待测水样中加入一定比例的细胞裂解液,细胞裂解后释放出ATP,在酶、氧气、ATP的参与下发生酶促反应,放出光子并产生固定波长的荧光,再利用荧光检测仪检测荧光信号获得相对光单位(relative light units,RLU)数值.利用已知的ATP校准曲线即可将RLU值转换成ATP浓度.该方法不需要生物培养过程,检测快、操作简便、成本较低[30],而且可以检测水样中不能培养的微生物,相较于HPC法具有明显的优势.但由于测得的ATP浓度难以直观地表达活菌数,需要通过转换因子--单位细胞的ATP量(ATP-per-cell)或单位细胞体积的ATP量(ATP-per-biovolume)换算得到待测水体中的生物量[31].而转换因子的确定方式复杂且尚存争议,这使得传统的ATP计数法在实验研究和常规检测中受到一定的限制.
2 基于流式细胞术改进的新兴评价方法流式细胞术是一种对快速直线流动状态的单列细胞或生物颗粒进行逐个、多参数、快速地定性、定量分析或分选的分析方法,具有检测速度快、测量参数多、采集数据量大、分选纯度高、分析全面、方法灵活等特点[32].流式细胞仪(flow cytometer,FCM)是以流式细胞术为核心技术发展的分析测试仪器,其应用于饮用水水质微生物检测使得快速测定水样中细菌数得以实现,可以用于测定水样总细胞浓度,还能计数活性细胞和抗体细胞[33].
流式细胞仪由液流系统、光学系统、信号收集与转换系统和分析系统组成.图 1为4参数流式细胞仪的工作原理[34].如图 1所示,染色后的细胞或颗粒悬浮液进入液流系统,在鞘液的包被下细胞或颗粒单行排列,依次进入检测区,聚焦激光束垂直照射样品流,产生散射光和激发荧光,被前向光电二极管(photodiode, PD)和侧向90°方向的光电倍增管(photomultiplier tube, PMT)接收.前向光电二极管获得前散射信号(forward light scatter, FSC),侧向光电倍增管获得侧散射信号(side light scatter, SSC)和荧光信号(FL1, FL3等).FSC能够反映细胞或颗粒的尺寸和折射率,SSC反映细胞内部结构,荧光信号则反映被标记细胞的生物特异性.测得不同参数的信号强度可用于区分各样品的亚种群,例如分析白血球样品时,散射光信号可以用来区分单核细胞、淋巴细胞和粒性白细胞,再通过荧光信号分析细胞表面的抗原进一步区分细胞[34].另外,不同染料与待测样品中细胞特异性结合后,可获得细胞总数、大小、活性、DNA含量等信息[9, 35].该方法测定水中细菌的检测范围为1×103~2×105 cells/mL,浓度超过该范围时对样品进行稀释即可[36].
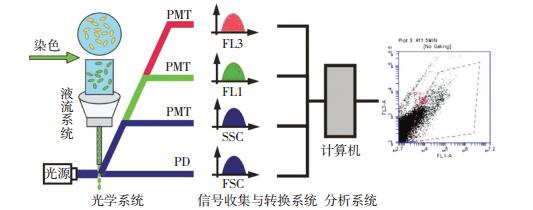
|
图 1 流式细胞仪基本原理 Figure 1 Basic principle of flow cytometer |
作为一个多功能的测试仪器,流式细胞仪应用于饮用水水质微生物检测方面具有极大的优势.基于流式细胞术改进的AOC、TCC、ATP评价方法较传统方法简单快速、准确度更高,受到越来越多研究者的关注,改进的TCC测定方法已被瑞士推广为细菌数检测的标准方法[12].
2.1 基于流式细胞术改进的AOC评价方法传统的AOC测定方法采用平板计数来测量水样培养前后的微生物量,操作繁琐且耗时较长.Hammes等[37]提出全新的AOC测定方法,如图 2所示,以天然菌作为接种液,在经0.22 μm滤膜过滤后的水样中培养至稳定期,利用流式细胞仪测定起始细菌浓度(cell conc. 1)和培养3 d后的细菌浓度(cell conc. 3),两者的差值即为细菌增长量,再通过统一的转换系数k来获得AOC值.该方法具有明显的优势,检测时间大大缩短,而且操作简单,不需要特殊的菌源.
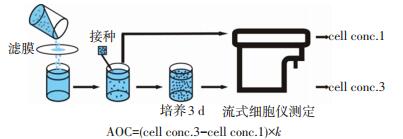
|
图 2 基于流式细胞术改进的AOC测定方法 Figure 2 Improved AOC measurement based on flow cytometry |
基于流式细胞术改进的TCC测定过程如图 3所示,水样用经0.22 μm滤膜过滤后的依云(Evian)水稀释,加入1%的染料(SYBR Green I),在30 ℃黑暗环境下培养15 min左右,再用流式细胞仪测定[38].
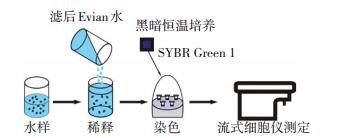
|
图 3 基于流式细胞术改进的TCC测定方法 Figure 3 Improved TCC measurement based on flow cytometry |
流式细胞术的引入极大地促进了TCC作为饮用水处理和管网系统中水质生物稳定性评价指标的推广.基于流式细胞术改进的TCC评价方法具有多方面的优势[27, 38]:1)全面,其检出数据比HPC高2个数量级,而且能检测不能培养的细菌;2)灵敏,FCM能够准确地检测出TCC低至5%的变化,相当于常规水源水体0.5~5 μg/L的有机碳消耗,且检出限低;3)稳定,测试结果重现性好;4)省时,与HPC测定时间至少需要24 h相比,流式细胞仪计数仅需15 min.
Hammes等[38]以瑞士某饮用水处理厂O3-GAC-UF工艺为研究对象,通过对各个工艺进出水取样检测,比较HPC、TCC、ATP 3个指标用于评价水处理工艺微生物状况的优劣性,认为基于流式细胞术改进的TCC评价方法不仅较大程度地缩短了检测时间,而且能检测出不能培养的细菌,较HPC、ATP指标具有显著的优势,这与Hoefel和Phe等[39-40]的研究结果一致.Lautenschlager等[27]对瑞士某大型管网系统中不同停留时间的管网点长期连续取样检测,利用多参数评价该管网系统水质生物稳定性.结果显示利用流式细胞仪测得的TCC数据可靠,不仅能够指示水质恶化,还可以佐证AOC、TOC及HPC数据.Siebel等[41]对实际给水管网取样,研究了基于流式细胞术改进方法测得的TCC、ATP及传统的HPC指标之间的相关性,发现ATP和TCC数据有较好的相关性,但二者与HPC的相关性均很弱.Liu等[42]的类似研究表明,TCC与ATP及HPC之间均没有较好的相关性,但当ATP质量浓度大于3 ng/L时,高核酸细菌(HNA bacteria)的浓度与ATP存在一定的线性相关性,这可能是由于TCC在检测较低浓度的生物量变化时较ATP更为灵敏.
TCC直接反映了水体中的微生物总量,对水质稳定性的评价具有重要意义.一些研究人员认为基于流式细胞法的TCC指标将会成为水处理过程及管网系统中水质的常规监测指标,同时可以作为某些水处理工艺的设计和优化参数[38-39, 43-45].然而,由于TCC还可能含有失去活性的细菌,不能充分地描述紫外消毒等工艺的效能.因此,将TCC与反映活性生物量的ATP或完整细胞浓度(intact cell concentration, ICC)结合起来用于评估水质微生物状况更具有指导意义[38, 41].
2.3 基于流式细胞术改进的ATP评价方法流式细胞仪的应用可较为准确且相对简单地确定ATP与细菌量的转化系数,克服了ATP计数法的限制.水样经碘化丙啶(PI)染色后可以用FCM快速测得完整细胞浓度ICC,从而计算出ATP-per-cell,利用FCM SSC数据可以估算微生物细胞体积,进而求得ATP-per-biovolume[31].
Hammes等[31]在评价水环境中天然微生物群落活性时,分别从湖泊、溪流、地下水、管网中取大量水样,利用基于流式细胞术改进的ATP评价方法,发现微生物细胞ATP浓度与ICC、完整细胞体积浓度相关性良好,但与HPC浓度没有相关性.并拟合得出平均的ATP-per-cell为1.75×10-10 nmol/cell,平均的ATP-per-cell为2.95×10-9 nmol/μm3.Velten等[46]利用基于流式细胞术改进的ATP评价方法分析饮用水颗粒活性炭上的生物相,得到ATP-per-cell值为(1.3~4.5)×10-10 nmol/cell,与Magic-Knezev等[47]利用传统的落射荧光显微技术与ATP计数法结合所测得的颗粒活性炭过滤器中细菌的ATP-per-cell (0.41×10-10 nmol/cell)数值结果基本一致.
基于流式细胞术改进的ATP评价方法不仅简化了转化系数的确定过程,而且提高了测量精度,解决了制约ATP计数法推广的主要难题.但其检出限比较高,在检测较低浓度的细菌量变化时没有TCC灵敏;同时对于低核酸细菌(LNA bacteria),由于其每个细胞所含的ATP数量很少,很难用ATP法检测其增长[27, 48].此外,ATP检出浓度还受细胞大小、细胞活性、细胞生存能力等影响[49].近年来,ATP计数法评价指标已开始用于饮用水、地下水、生物滤池、管网生长环等水环境领域的生物量检测[31].有研究人员认为[9, 50],基于流式细胞术改进的ATP评价方法可以作为饮用水常规监测手段,ATP评价指标可以用于监测饮用水工艺过程中微生物活性的改变.
3 基于分子生物学技术的新兴评价方法测定饮用水处理工艺或管网系统中水体的AOC、TCC、ATP等指标能获得微生物生长潜能、细菌数量、活性等信息从而评价水质变化,但不能分析水质变化的根本原因.基于分子生物学技术的变性梯度凝胶电泳(denaturing gradient gel electrophoresis, DGGE)和焦磷酸测序(pyrosequencing)等新兴评价方法可以根据水环境中生物群落的多样性、种属及其丰度等信息,快速确定水质细菌学变化,并从微生物生态学角度分析水质变化的根本原因,更深入地评价水质安全性和生物稳定性.
3.1 变性梯度凝胶电泳法变性梯度凝胶电泳(DGGE)可以通过分离DNA片段来获得微生物群落指纹信息,是目前应用最为广泛的DNA指纹技术.其基本原理是长度相同而碱基组成不同的微生物DNA序列在线性梯度浓度的DNA变性剂聚丙烯酰胺凝胶中发生解链,序列不同的DNA片段解链行为不同而导致其电泳迁移率不同[51],从而在凝胶上形成一系列电泳条带,条带的数量对应于微生物群落中优势菌群的数量,条带的多寡、亮度和位置则可以用来监测微生物群落的改变,半定量地估计微生物种属丰度.
基于聚合酶链式反应(polymerase chain reaction, PCR)扩增技术的DGGE (PCR-DGGE)由于可以通过直接的DNA提取获得水样中微生物群落指纹而被广泛应用.测试过程[52]如图 4所示,提取水样中微生物DNA,得到不同菌群的DNA混合物;以该混合物为模板,与具有特异性的DNA引物对进行PCR扩增,得到长度相同而碱基组成不同的各生物菌群DNA扩增子;通过DGGE技术将DNA扩增子分开,得到一系列电泳条带,即为待测水样的微生物群落指纹;将电泳条带割胶纯化,在PCR扩增后进行测序,获得微生物具体种属信息[53].
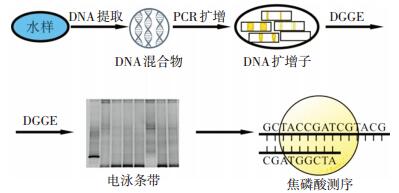
|
图 4 PCR-DGGE测定流程示意 Figure 4 Process of PCR-DGGE measurement |
DGGE可以实现多样品同时测定,并快速确定微生物群落结构变化,深入分析饮用水生物稳定性的变化及其根本原因.Sekar等[54]利用HPC和DGGE方法对比评价实际管网的水质生物稳定性,发现DGGE更能反映管网水质随水力条件的变化.DGGE还可分析饮用水嗅味变化、评价不同给水处理工艺出水的细菌学水质,并被推荐用于描述和评价饮用水水质变化[55-57].
3.2 焦磷酸测序焦磷酸测序(Pyrosequencing)方法基于边合成边测序原理,通过磷酸法产生的光学信号分析获得碱基信息,最终得到待测水样中微生物的DNA序列.与已知DNA序列库对照,可获得微生物种属及其丰度信息.其利用DNA聚合酶、ATP硫酸化酶、荧光素酶和双磷酸酶的协同作用,将PCR反应中每一个碱基的延伸与一次荧光信号的释放偶联起来,通过记录荧光信号的有无和强度,达到实时测定DNA序列的目的.
利用焦磷酸测序法检测水环境样本,可以通过分析群落结构变化来判断水质的生物稳定性,同时根据优势菌属信息确定引起管网水微生物再生的主要因素,还能够利用获得的细菌种类及丰度信息来分析水质恶化的根源,以提出相应的水质优化方案.
Defalont等[58]利用焦磷酸测序分析管网水体中微生物的相互关系,发现87.6%的变形虫都负载了较多数量的潜在病原体分支杆菌.Pinto等[59]利用焦磷酸测序分析水源水质及水处理单元对管网细菌群落结构的影响,发现快速砂滤对管网水环境细菌的群落结构起主要作用,可以利用过滤单元的微环境来控制微生物种类和数量,保证管网水质生物稳定性.焦磷酸测序还可与DGGE指纹技术联合用于评价饮用水水质生物稳定性.Lautenschlager等[27]利用DGGE和焦磷酸测序结合分析实际管网的水质变化,得出停留时间较长的管网水样水质发生变化的根本原因是丛毛单胞菌属的增加;同时发现DGGE和焦磷酸测序的分析结果与基于流式细胞术改进的TCC数据可以相互佐证,并认为这两个指标可作为评价水质生物稳定性的补充指标.
焦磷酸测序法能够检测丰度<1%的菌群,可提供微生物种属及丰度信息,测试结果可靠,同时,其测试过程快速且操作简单,是目前应用最广泛的第二代测序平台.近年来,具有更高性价比的Illumina测序平台受到研究者的关注,并有逐渐替代焦磷酸测序的潜能[12, 60].
微生物群落分析专业性强,成本较高,在评价饮用水水质生物稳定性方面的应用还较少,但是基于分子生物学技术的新兴生物稳定性评价方法可以深入地了解水处理过程及管网输配过程水环境中微生物组成变化,对建立更完善的饮用水生物稳定性评价指标体系具有重要意义,为从根本上探究水质生物稳定性指明了方向.
4 展望传统的水质生物稳定性评价方法检测成本低、操作简单,为我国相关标准推荐的方法,但其存在准确性差、分析结果局限等缺点;基于流式细胞术改进的评价方法检测耗时少,测定结果更为可靠,有望在水质监测和工程应用中推广.基于分子生物学的评价方法能够准确获得水环境中微生物群落信息,从微生物生态学角度分析水质变化的根本原因,但其仪器及检测成本均较高,目前在水质检测方面应用还较少.随着DNA测序成本的逐渐降低以及自动化程度的提高,高通量测序有望成为水质分析的辅助工具,为饮用水水质安全提供有力的保障.
| [1] | KOOIJ VAN DER D. Biological stability: A multidimensional quality aspect of treated water[J]. Water, Air, and Soil Pollution, 2000, 123 : 25-34. DOI: 10.1023/A:1005288720291 |
| [2] | VITAL M, FVCHSLIN H P, HAMMES F, et al. Growth of Vibrio cholerae O1 Ogawa Eltor in freshwater[J]. Microbiology, 2007, 153 (7) : 1993-2001. DOI: 10.1099/mic.0.2006/005173-0 |
| [3] | VITAL M, DIGNUM M, MAGIC-KNEZEV A, et al. Flow cytometry and adenosine tri-phosphate analysis: Alternative possibilities to evaluate major bacteriological changes in drinking water treatment and distribution systems[J]. Water Research, 2012, 46 (15) : 4665-4676. DOI: 10.1016/j.watres.2012.06.010 |
| [4] | FRANCISQUE A, RODRIGUEZ M J, MIRANDA-MORENO L F, et al. Modeling of heterotrophic bacteria counts in a water distribution system[J]. Water Research, 2009, 43 (4) : 1075-1087. DOI: 10.1016/j.watres.2008.11.030 |
| [5] | BARTRAM J, COTRUVO J, EXNER M, et al. Heterotrophic plate count measurement in drinking water safety management[J]. International Journal of Food Microbiology, 2004, 92 (3) : 241-247. DOI: 10.1016/j.ijfoodmicro.2003.08.005 |
| [6] | CARTER J T, RICE E W, BUCHBERGER S G, et al. Relationships between levels of heterotrophic bacteria and water quality parameters in a drinking water distribution system[J]. Water Research, 2000, 34 (5) : 1495-1502. DOI: 10.1016/S0043-1354(99)00310-3 |
| [7] | ALLEN M J, EDBERG S C, REASONER D J. Heterotrophic plate count bacteria-what is their significance in drinking water?[J]. International Journal of Food Microbiology, 2004, 92 (3) : 265-274. DOI: 10.1016/j.ijfoodmicro.2003.08.017 |
| [8] | REASONER D J. Heterotrophic plate count methodology in the United States[J]. International Journal of Food Microbiology, 2004, 92 (3) : 307-315. DOI: 10.1016/j.ijfoodmicro.2003.08.008 |
| [9] | BERNEY M, VITAL M, HVLSHOFF I, et al. Rapid, cultivation-independent assessment of microbial viability in drinking water[J]. Water Research, 2008, 42 (14) : 4010-4018. DOI: 10.1016/j.watres.2008.07.017 |
| [10] | KOOIJ VAN DER D, VROUWENVELDER J S, VEENENDAAL H R. Elucidation and control of biofilm formation processes in water treatment and distribution using the unified biofilm approach[J]. Water Science and Technology, 2003, 47 (5) : 83-90. |
| [11] | BURTSCHER M M, ZIBUSCHKA F, MACH R L, et al. Heterotrophic plate count vs.in situ bacterial 16S rRNA gene amplicon profiles from drinking water reveal completely different communities with distinct spatial and temporal allocations in a distribution net[J]. Water Sa, 2009, 35 (4) : 495-504. |
| [12] | DOUTERELO I, BOXALL J B, DEINES P, et al. Methodological approaches for studying the microbial ecology of drinking water distribution systems[J]. Water Research, 2014, 65 : 134-156. DOI: 10.1016/j.watres.2014.07.008 |
| [13] | TRULLEYOVÁ Š, RUL1K M. Determination of biodegradable dissolved organic carbon in waters: Comparison of batch methods[J]. Science of the Total Environment, 2004, 332 (1/2/3) : 253-260. |
| [14] | SERVAIS P, BILLEN G, HASCOËT M. Determination of the biodegradable fraction of dissolved organic matter in waters[J]. Water Research, 1987, 21 (4) : 445-450. DOI: 10.1016/0043-1354(87)90192-8 |
| [15] | SERVAIS P, ANZIL A, VENTRESQUE C. Simple method for determination of biodegradable dissolved organic-carbon in water[J]. Applied and Environmental Microbiology, 1989, 55 (10) : 2732-2734. |
| [16] | VOLK C, RENNER C, ROBERT C, et al. Comparison of two techniques for measuring biodegradable dissolved organic carbon in water[J]. Environmental Technology, 1994, 15 (6) : 545-556. DOI: 10.1080/09593339409385460 |
| [17] | KOOIJ VAN DER D, VISSER A, ORANJE J P. Multiplication of fluorescent pseudomonads at low substrate concentrations in tap water[J]. Antonie Van Leeuwenhoek, 1982, 48 (3) : 229-243. DOI: 10.1007/BF00400383 |
| [18] | KAPLAN L A, NEWBOLD J D. Measurement of streamwater biodegradable dissolved organic carbon with a plug-flow bioreactor[J]. Water Research, 1995, 29 (12) : 2696-2706. DOI: 10.1016/0043-1354(95)00135-8 |
| [19] | ESCOBAR I C, RANDALL A A. Assimilable organic carbon (AOC) and biodegradable dissolved organic carbon (BDOC): Complementary measurements[J]. Water Research, 2001, 35 (18) : 4444-4454. DOI: 10.1016/S0043-1354(01)00173-7 |
| [20] | LIU Wenjun, WU Hongwei, WANG Zhansheng, et al. Investigation of assimilable organic carbon (AOC) and bacterial regrowth in drinking water distribution system[J]. Water Research, 2002, 36 (4) : 891-898. DOI: 10.1016/S0043-1354(01)00296-2 |
| [21] | LECHEVALLIER M W, WELCH N J, SMITH D B. Full-scale studies of factors related to coliform regrowth in drinking water[J]. Applied and Environmental Microbiology, 1996, 62 (7) : 2201-2211. |
| [22] | KOOIJ VAN DER D. Characterization and classification of fluorescent pseudomonads isolated from tap water and surface water[J]. Antonie Van Leeuwenhoek, 1979, 45 (2) : 225-240. DOI: 10.1007/BF00418586 |
| [23] | KOOIJ VAN DER D. The occurrence of Pseudomonas spp. in surface water and in tap water as determined on citrate media[J]. Antonie Van Leeuwenhoek, 1977, 43 (2) : 187-197. DOI: 10.1007/BF00395673 |
| [24] | LECHEVALLIER M W, SHAW N E, KAPLAN L A, et al. Development of a rapid assimilable organic-carbon method for water[J]. Applied and Environmental Microbiology, 1993, 59 (5) : 1526-1531. |
| [25] | VELTEN S, BOLLER M, KÖSTER O, et al. Development of biomass in a drinking water granular active carbon (GAC) filter[J]. Water Research, 2011, 45 (19) : 6347-6354. DOI: 10.1016/j.watres.2011.09.017 |
| [26] | HAMMES F, BERGER C, KÖSTER O, et al. Assessing biological stability of drinking water without disinfectant residuals in a full-scale water supply system[J]. Journal of Water Supply: Research and Technology-AQUA, 2010, 59 (1) : 31. DOI: 10.2166/aqua.2010.052 |
| [27] | LAUTENSCHLAGER K, HWANG C, LIU W, et al. A microbiology-based multi-parametric approach towards assessing biological stability in drinking water distribution networks[J]. Water Research, 2013, 47 (9) : 3015-3025. DOI: 10.1016/j.watres.2013.03.002 |
| [28] | RINTA-KANTO J M, LEHTOLA M J, VARTIAINEN T, et al. Rapid enumeration of virus-like particles in drinking water samples using SYBR green I-staining[J]. Water Research, 2004, 38 (10) : 2614-2618. DOI: 10.1016/j.watres.2004.03.008 |
| [29] |
梁涛.可生物同化有机碳在给水处理单元过程中的变化规律[D].哈尔滨:哈尔滨工业大学, 2009.
LIANG Tao. Variation of assimilable organic carbon in drinking water treatment processes[D]. Harbin:Harbin Institute of Technology, 2010. |
| [30] | DELAHAYE E, WELTÉ B, LEVI Y, et al. An ATP-based method for monitoring the microbiological drinking water quality in a distribution network[J]. Water Research, 2003, 37 (15) : 3689-3696. DOI: 10.1016/S0043-1354(03)00288-4 |
| [31] | HAMMES F, GOLDSCHMIDT F, VITAL M, et al. Measurement and interpretation of microbial adenosine tri-phosphate (ATP) in aquatic environments[J]. Water Research, 2010, 44 (13) : 3915-3923. DOI: 10.1016/j.watres.2010.04.015 |
| [32] | WATSON D A, BROWN L O, GASKILL D R, et al. A flow cytometer for the measurement of Raman spectra[J]. Cytometry Part A, 2008, 73A (2) : 119-128. DOI: 10.1002/(ISSN)1552-4930 |
| [33] | TUNG Yichung, ZHANG Ming, LIN Chihting, et al. PDMS-based opto-fluidic micro flow cytometer with two-color, multi-angle fluorescence detection capability using PIN photodiodes[J]. Sensors and Actuators B: Chemical, 2004, 98 (2/3) : 356-367. |
| [34] | CHO S H, GODIN J M, CHEN Chunhao, et al. Review article: Recent advancements in optofluidic flow cytometer[J]. Biomicrofluidics, 2010, 4 (4) : 43001. DOI: 10.1063/1.3511706 |
| [35] | GAWAD S, SCHILD L, RENAUD P. Micromachined impedance spectroscopy flow cytometer for cell analysis and particle sizing[J]. Lab On a Chip, 2001, 1 (1) : 76. DOI: 10.1039/b103933b |
| [36] |
文刚, 王静怡, 黄廷林, 等. 流式细胞仪在水处理中的应用现状与展望[J].
中国给水排水, 2014, 30 (18) : 58-62.
WEN Gang, WANG Jingyi, HUANG Tinglin, et al. Application of flow cytometer to water treatment: status and perspectives[J]. China Water & Wastewater, 2014, 30 (18) : 58-62. |
| [37] | HAMMES F A, EGLI T. New method for assimilable organic carbon determination using flow-cytometric enumeration and a natural microbial consortium as inoculum[J]. Environmental Science & Technology, 2005, 39 (9) : 3289-3294. |
| [38] | HAMMES F, BERNEY M, WANG Yingying, et al. Flow-cytometric total bacterial cell counts as a descriptive microbiological parameter for drinking water treatment processes[J]. Water Research, 2008, 42 (1/2) : 269-277. |
| [39] | HOEFEL D, GROOBY W L, MONIS P T, et al. Enumeration of water-borne bacteria using viability assays and flow cytometry: A comparison to culture-based techniques[J]. Journal of Microbiological Methods, 2003, 55 (3) : 585-597. DOI: 10.1016/S0167-7012(03)00201-X |
| [40] | PHE M, DOSSOT M, GUILLOTEAU H, et al. Nucleic acid fluorochromes and flow cytometry prove useful in assessing the effect of chlorination on drinking water bacteria[J]. Water Research, 2005, 39 (15) : 3618-3628. DOI: 10.1016/j.watres.2005.06.002 |
| [41] | SIEBEL E, WANG Yingying, EGLI T, et al. Correlations between total cell concentration, total adenosine tri-phosphate concentration and heterotrophic plate counts during microbial monitoring of drinking water[J]. Drinking Water Engineering and Science Discussions, 2008, 1 (1) : 71-86. DOI: 10.5194/dwesd-1-71-2008 |
| [42] | LIU Gang, MARK van der E J, VERBERK J Q J C, et al. Flow cytometry total cell counts: A field study assessing microbiological water quality and growth in unchlorinated drinking water distribution systems[J]. BioMed Research International, 2013, 2013 : 1-10. |
| [43] | WANG Yingying, HAMMES F, ROY DE K, et al. Past, present and future applications of flow cytometry in aquatic microbiology[J]. Trends in Biotechnology, 2010, 28 (8) : 416-424. DOI: 10.1016/j.tibtech.2010.04.006 |
| [44] | HO L, BRAUN K, FABRIS R, et al. Comparison of drinking water treatment process streams for optimal bacteriological water quality[J]. Water Research, 2012, 46 (12) : 3934-3942. DOI: 10.1016/j.watres.2012.04.041 |
| [45] | PREST E I, HAMMES F, KÖTZSCH S, et al. Monitoring microbiological changes in drinking water systems using a fast and reproducible flow cytometric method[J]. Water Research, 2013, 47 (19) : 7131-7142. DOI: 10.1016/j.watres.2013.07.051 |
| [46] | VELTEN S, HAMMES F, BOLLER M, et al. Rapid and direct estimation of active biomass on granular activated carbon through adenosine tri-phosphate (ATP) determination[J]. Water Research, 2007, 41 (9) : 1973-1983. DOI: 10.1016/j.watres.2007.01.021 |
| [47] | MAGIC-KNEZEV A, KOOIJ VAN DER D. Optimisation and significance of ATP analysis for measuring active biomass in granular activated carbon filters used in water treatment[J]. Water Research, 2004, 38 (18) : 3971-3979. DOI: 10.1016/j.watres.2004.06.017 |
| [48] | WANG Yingying, HAMMES F, BOON N, et al. Isolation and characterization of low nucleic acid (LNA)-content bacteria[J]. Isme Journal, 2009, 3 (8) : 889-902. DOI: 10.1038/ismej.2009.46 |
| [49] | KARL D M. Cellular nucleotide measurements and applications in microbial ecology[J]. Microbiological Reviews, 1980, 44 (4) : 739-796. |
| [50] | WIELEN VAN DER P W J J, KOOIJ VAN DER D. Effect of water composition, distance and season on the adenosine triphosphate concentration in unchlorinated drinking water in the Netherlands[J]. Water Research, 2010, 44 (17) : 4860-4867. DOI: 10.1016/j.watres.2010.07.016 |
| [51] | MUYZER G, SMALLA K. Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology[J]. Antonie Van Leeuwenhoek, 1998, 73 (1) : 127-141. DOI: 10.1023/A:1000669317571 |
| [52] | ERCOLINI D. PCR-DGGE fingerprinting: Novel strategies for detection of microbes in food[J]. Journal of Microbiological Methods, 2004, 56 (3) : 297-314. DOI: 10.1016/j.mimet.2003.11.006 |
| [53] | DOUTERELO I, BOXALL J B, DEINES P, et al. Methodological approaches for studying the microbial ecology of drinking water distribution systems[J]. Water Research, 2014, 65 : 134-156. DOI: 10.1016/j.watres.2014.07.008 |
| [54] | SEKAR R, DEINES P, MACHELL J, et al. Bacterial water quality and network hydraulic characteristics: A field study of a small, looped water distribution system using culture-independent molecular methods[J]. Journal of Applied Microbiology, 2012, 112 (6) : 1220-1234. DOI: 10.1111/jam.2012.112.issue-6 |
| [55] | OTTERHOLT E, CHARNOCK C. Identification and phylogeny of the small eukaryote population of raw and drinking waters[J]. Water Research, 2011, 45 (8) : 2527-2538. DOI: 10.1016/j.watres.2011.02.008 |
| [56] | HO L, BRAUN K, FABRIS R, et al. Comparison of drinking water treatment process streams for optimal bacteriological water quality[J]. Water Research, 2012, 46 (12) : 3934-3942. DOI: 10.1016/j.watres.2012.04.041 |
| [57] | LI Zonglai, HOBSON P, WEI An, et al. Earthy odor compounds production and loss in three cyanobacterial cultures[J]. Water Research, 2012, 46 (16) : 5165-5173. DOI: 10.1016/j.watres.2012.06.008 |
| [58] | DELAFONT V, MOUGARI F, CAMBAU E, et al. First evidence of amoebae-mycobacteria association in drinking water network[J]. Environmental Science & Technology, 2014, 48 (20) : 11872-11882. |
| [59] | PINTO A J, XI Chuanwu, RASKIN L. Bacterial community structure in the drinking water microbiome is governed by filtration processes[J]. Environmental Science & Technology, 2012, 46 (16) : 8851-8859. |
| [60] | LUO Chengwei, TSEMENTZI D, KYRPIDES N, et al. Direct comparisons of illumina vs. roche 454 sequencing technologies on the same microbial community DNA sample[J]. Plos One, 2012, 7 : e300872. |
 2017, Vol. 49
2017, Vol. 49


