2. 北京建筑大学电气与信息工程学院, 北京 100044;
3. 建筑大数据智能处理方法研究北京市重点实验室, 北京 100044
2. School of Electrical and Information Engineering, Beijing University of Civil Engineering and Architecture, Beijing 100044;
3. Beijing Key Laboratory of Intelligent Processing for Building Big Data, Beijing 100044
水稻作为农作物研究的重要模式生物[1], 相关的基因组信息[2-3]、转录组数据积累得越来越多, 相关的研究在逐年增长.充足的数据使得人们能够对水稻基因的表达模式进行详细的分析.在过去的十多年里, 人们使用统计学方法对水稻基因的表达模式进行大量的研究[4-7], 包括组织转录特征[8]、水稻在外部胁迫下的转录响应[9]、田间环境表达模式[10]等.研究人员也整合相关资源构建了便于检索的集成数据库[11-14], 包括集成功能注释与基因组信息的OryzaBase[11]和RiceDB[12], 综合的基因共表达网络ATTED-Ⅱ[13], 功能相关网络RiceNetV2[14].Chandran和Jung对水稻相关的系统生物学的资源信息进行汇总, 讨论了水稻组学数据的发展过程以及最近的进展[15].
与模式植物拟南芥相比, 高等植物水稻的生长环境复杂, 培育时间长, 生物实验代价高昂.使得水稻的研究主要集中在基因层面整体转录模式的分析, 缺少蛋白质相互作用方面的研究.目前用于预测新的蛋白质相互作用的计算方法包括基于蛋白质空间结构的方法[16], 集成功能基因组信息的机器学习方法[17-18]以及基于进化保守性的同源映射方法[19-21].已知空间结构信息较少和计算效率低下的问题使得基于空间结构的方法并不适用于大规模植物蛋白质相互作用网络预测.与此同时, 功能注释信息的缺乏和已知的相互作用较少使得基于SVM等的机器学习方法也不适用于水稻蛋白质相互作用的预测.
基于进化保守性的同源预测方法不需要功能注释信息和金标准数据, 是目前研究的热点.在2011年和2012年, Chen的团队和Matt的团队基于同源映射方法分别预测了水稻的蛋白质相互作用网络, 并使用收集的数据以及预测的亚细胞定位信息对计算的蛋白质相互作用进行了分析说明[22-23].这两项研究包含5 573个水稻基因, 约占水稻基因总数14%.两项包含的基因重叠程度较高, 但是预测得到的相互作用差异较大, 重叠程度较低, Chen的工作PRIN中只有30%的相互作用在Matt的预测结果中出现.而且, Chen和Matt的工作是从整体上对水稻蛋白质相互作用的预测, 并不能够反应蛋白质相互作用的组织特异性.此外, 这两项工作完成时间较早, 对于后续出现大量的相互作用资源利用不够充分.因此, 水稻在基因和蛋白质的组织特异性表达与相互作用方面的研究十分滞后.
综上所述, 当前水稻组织特异性蛋白质相互作用的研究中存在如下几点问题:1)数据利用不充分, 组织特异基因的识别没有统一的标准; 2)目前的蛋白质相互作用网络的预测方法单一, 数据滞后, 网络覆盖程度不够; 3)缺少组织特异性蛋白质相互作用状况的研究.因此, 水稻组织特异性的蛋白质相互作用的预测依旧是一个挑战, 有待进一步的研究.
为了解决上述问题, 本文以水稻为研究对象提出一种组织特异性蛋白质相互作用网络构建方法, 并构建了水稻的组织特异性网络(PTSN4R).其目的是为预测组织特异的蛋白质相互作用提供一个合理的框架, 为研究人员从组织特异的角度来研究基因的表达和蛋白质相互作用提供便利.首先, 通过分析目前组织特异基因的识别方法确定了一致的识别标准, 进而识别出水稻各组织中组织特异基因, 并使用预测的水稻必需基因进行交叉验证.然后, 提出基于支持得分的同源映射方法并构建了水稻全基因组的蛋白质相互作用网络.最后, 根据识别的组织特异基因和预测的蛋白质相互网络构建了水稻各组织中组织特异的蛋白质相互作用网络, 并根据基因表达相关性筛选出了高可靠的蛋白质相互作用.
1 组织特异性蛋白质相互作用网络构建方法 1.1 组织特异基因的识别基因的组织特异性表达是基因重要特性之一.完成组织特异表达基因的识别需要根据样本表达特征将数据按照合适的层次划分到不同组织中.首先按照原始数据集中标注的组织划分, 然后根据样本间的表达相关性, 对相同以及相近组织的表达谱样本进行确认, 根据相似度合并成为该组织的表达数据集.不同品种的往往具有不同的表达特性, 因此, 将水稻数据集GSE19024中MH, ZS两个品种区分处理.
根据Nadezda与Marc对众多组织特异性表达指标的比较研究, 使用由Yanai等提出Tau[24]作为表达特异性的指标.
Tau的计算公式为
| $ \tau = \frac{{\sum\nolimits_{i = 1}^n {\left( {1-{{\bar x}_i}} \right)} }}{{n-1}};\;{{\bar x}_i} = \frac{{{x_i}}}{{\mathop {{\rm{max}}}\limits_{1 \le i \le n} ({x_i})}}. $ | (1) |
式中:xi是基因x在第i个组织中的表达值, xi 是使用最大分量值正则化的表达值, n是组织的个数.根据公式可看出, Tau值越接近于1则表示这个基因是组织特异表达的.
同时, 使用基于同源性方法预测了水稻的必需基因, 用来与识别的差异表达基因进行对比验证.a)与DEG数据库的最佳比对必须满足E-value <1e-10, Identity>40%;b)与D-Non-EG数据库的最佳比对结果必须满足E-value <1e-10, Identity <40%.
1.2 直系同源基因的预测方法根据目前蛋白质相互作用网络的完善程度选择6个参考物种(大肠杆菌, 酵母菌, 蠕虫, 果蝇, 人类, 拟南芥)作为源物种.使用局部序列比对搜索工具(BLAST)来识别目标物种基因在参考物种中的直系同源基因.首先, 将水稻以及6个参考物种的蛋白质序列制作成BLASTP程序可用的蛋白质序列局部比对数据库.然后, 使用BLASTP程序在6个物种的蛋白质序列数据库中进行BLAST比对搜索水稻基因的同源基因.计算水稻所有蛋白质的直系同源蛋白质序列以及每对同源基因对应的E值(E-value)和一致性(Identity)和比特得分(Bit Score).满足E-value>1e-10的序列认为与目标序列存在直系同源关系.
1.3 直系同源映射和蛋白质相互作用评分现有直系同源映射方法均从源物种出发, 搜索已有相互作用(A′, B′)在目标物种中的直系同源对作为预测的蛋白质相互作用.这一策略不能确定预测的相互作用关系受到已有相互作用关系的支持程度.为了解决这一问题, 提出了一种新的基于直系同源映射原理的方法来预测蛋白质相互作用.
基于支持得分的同源映射方法步骤, 见图 1.其中, 图 1(a)为主要的步骤说明, 图 1(b)为直系同源基因识别以及直系同源映射的示意图.首先根据直系同源基因预测方法分别识别出A, B的直系同源的所有基因, 存入与B′.然后检查所有可能A′i与B′j在源物种中是否存在实验验证的相互作用关系.如果A′i与B′j存在相互作用, 而且同时满足JE <1e-40, JI <0.7.那么就认为 <A′i, B′j>就是支持 <A, B>存在相互作用的PPI.根据式(5)计算 <A, B>的支持得分.如果在源物种中发现多对满足直系同源映射的相互作用关系, 见图 1(b), 那么认为这一相互作用更为可靠.基于同源映射方法来预测相互作用时往往会集成多个物种的相互作用数据.
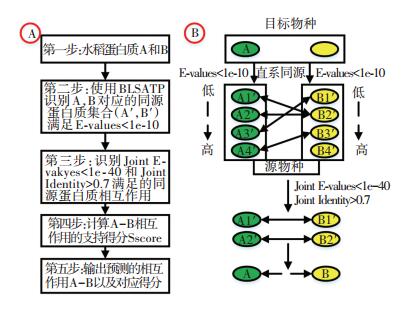
|
图 1 蛋白质相互作用预测及评分示意 Figure 1 Diagram of the prediction and evaluation of PPI |
通常使用联合序列相似性作为直系同源映射可靠程度评价标准.联合序列比对一致性JI, 与联合E值JE是常用的评价指标, 定义如下:
| $ {J_I} = \sqrt {{I_A}*{I_B}} . $ | (2) |
| $ {J_E} = \sqrt {{E_A}*{E_B}} . $ | (3) |
式中:IA, IB分别为蛋白质A, B的比对一致性值; EA, EB分别为蛋白质A, B的E值.JI, JE作为阈值筛选相互作用只能反应最佳匹配的同源映射对于预测的蛋白质相互作用的影响.不能正确的评价多个匹配对于预测出的相互作用的影响.为评价预测的蛋白质相互作用受到多个匹配的影响程度, 提出了支持得分Sscore来衡量预测的相互作用被源物种中的相互作用的支持程度.比特得分反映两序列的匹配程度, 本文使用正规化的比特得分来构建支持得分Sscore.计算Sscore的基本步骤如下:
1) 将A, B的直系同源基因的比特得分分别使用本组得分最大值正规化.
| $ {{\hat a}_i} = \frac{{{a_i}}}{{\mathop {{\rm{max}}}\limits_{1 \le i \le n} ({a_i})}};{{\mathit{\hat b}}_j} = \frac{{{b_j}}}{{\mathop {{\rm{max}}}\limits_{1 \le i \le m} ({b_j})}}. $ | (4) |
式中:ai为A的第i个同源蛋白质A′i的比特得分.bj为B的第j个同源蛋白质B′j的比特得分.n为源物种中与目标物种蛋白质A存在直系同源关系的蛋白质的数目.m为源物种中与目标物种蛋白质B存在直系同源关系的蛋白质的数目.
2) 那么支持得分Sscore为两个直系同源组中所有实验验证的相互作用的几何平均值的和.
| $ {S_{{\rm{score}}}} = \sum\limits_{ < {{\mathit{A'}}_i}-{{B'}_j} > \in Is} {\sqrt {{{\hat a}_i}*{{\mathit{\hat b}}_j}} } . $ | (5) |
式中:Is为物种中已经验证的所有的蛋白质相互作用的集合.
1.4 组织特异的蛋白质相互作用提取从全基因组层面计算得到了蛋白质相互作用网络与具体的组织或者发育过程无关.如果某一组织中相互作用对的一个蛋白质不存在, 那么这一相互作用就不会发生.针对每个特定组织, 抽取特异表达的基因来构建组织特异的子网, 并依据共表达相关性构建高可靠的组织特异的蛋白质相互作用网络.具体构建方法如下:
算法1:组织特异性蛋白质相互作用提取算法
输入:全部基因集Gall, 组织特异表达基因集Gtis, 完整蛋白质相互作用网Pall
输出:特定组织的子网Ptis, 高可靠的相互作用子网Phc
k←size(Gtis); n←size(Gall); Ptis←∅
FOR 每个基因 gi ∈Gtis
FOR 每个基因gj∈Gall
IF (gi≠gj)
t←PCC(gi, gj)
ELSE
t←-1
END
IF (<gi, gj>∈Pall)
Ptis←Ptis∪ <gi, gj>
END
IF (t≥0.7&&<gi, gj>∈Pall)
Phc←Phc∪ <gi, gj>
END
END
END
通过分析算法伪代码可知算法1包含两重循环, 因此算法1的时间复杂度为O(n2).
使用NCBI Gene ID作为统一的标识, 使得所有节点可以直接链接到NCBI数据库, 方便用户获取基因的注释信息并转向相关资源进行进一步分析.
1.5 组织特异的蛋白质相互网络构建整体方法提出的组织特异性蛋白质相互作用网络构建方法见图 2.构建过程包括3个主要步骤:1)识别与验证组织特异表达的基因; 2)基于同源映射方法预测全基因组蛋白质相互作用; 3)构建组织特异基因的蛋白质相互作用子网, 并筛选可靠性较高的蛋白质相互作用.图 2中蓝色框线标识的部分分别为上述主要步骤的结果, 分别为预测的组织特异的基因, 组织特异基因的共表达网络, 预测的全基因组蛋白质相互作用网络, 预测的组织特异的蛋白质相互作用子网.
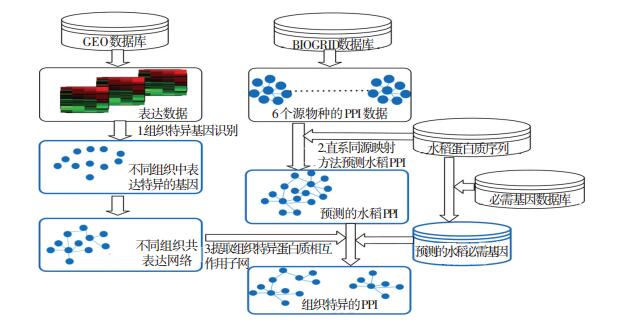
|
图 2 水稻组织特异性蛋白质相互作用网络计算方法 Figure 2 Tissue-specific protein-interaction network construction method for rice |
根据上述方法构建了水稻的组织特异的蛋白质相互作用子网(PTSN4R).图 2中蓝色框标识的部分提供下载.
2.1 数据来源 2.1.1 表达数据从GEO数据库收集了3个平台的5组公开的水稻表达谱数据, 详见表 1.
| 表 1 使用的水稻表达数据集 Table 1 Used expression data of rice |
开放获取的蛋白质相互作用数据库BIOGRID作为模式生物相互作用数据的来源.从中收集整理了6个参考物种(大肠杆菌Escherichia coli, 酵母菌Saccharomyces cerevisiae, 蠕虫Caenorhabditis elegans, 果蝇Drosophila melanogaster, 人类Homo sapiens, 拟南芥Arabidopsis thaliana)的数据作为同源性映射方法中源物种的相互作用, 见表 2.
| 表 2 6个参考物种相互作用网络统计 Table 2 The number of interactions in six reference species |
水稻(粳稻, Oryza sativa subsp.japonica)以及6个参考物种的蛋白质序列数据从NCBI参考基因组数据库获得.必需基因的蛋白质序列数据从必需基因数据库DEG[25]收集.
2.2 水稻组织特异基因的识别结果在五组数据上分别进行组织特异表达基因的识别.由于同一数据集中相似度较高的样本会降低特异表达识别的准确度, 在数据预处理阶段将相关系数大于90%的样本合并为一组.各个数据集上基因的Tau值的分布见图 3.从图 3可看出Tau值在靠近0.8的位置出现单峰.根据Tau值的分布以及参考文献[26], 选取Tau>0.8的基因作为组织特异的基因.GSE13161的生物样本为不同的细胞类型, 先按照各个细胞类型进行组织特异基因的识别, 然后将各类型细胞的识别结果归并到对应的组织中.
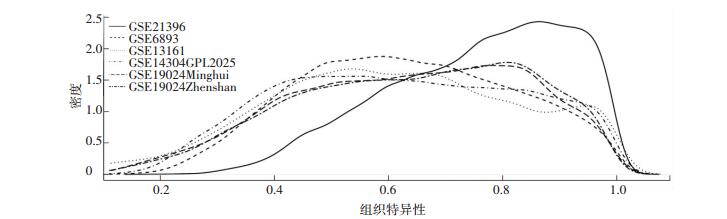
|
图 3 各个数据集上Tau值的分布 Figure 3 The distribution of Tau on each dataset |
此时得到水稻23个不同的组织的特异表达基因.各个数据集中检测到的特异表达基因数目并不一致, 这些差异同时也表明当前的数据数量、质量和计算方法不能够提供组织特异表达的完全确定的证据.人们对于特异表达的研究还需要进一步的细化.
数据集GSE14304, GSE19024, GSE21396对应的文献中, 作者提供了部分组织特异的基因.图 4展示了识别的组织特异的基因与对应参考文献识别结果的重叠程度.从图 4可看出使用Tau指标识别的组织特异基因基本包含参考文献给出的基因, 并且Tau方法识别了更多的组织特异的基因[26].
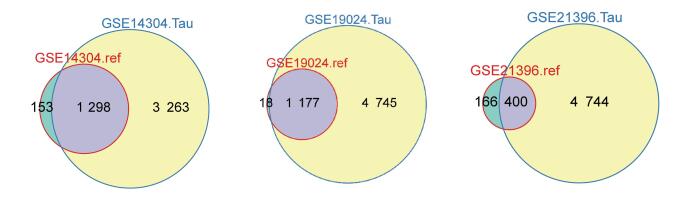
|
图 4 基于Tau识别的组织特异基因与参考文献比对结果 Figure 4 Comparison of tissue-specific genes in ref papers and by Tau method |
根据必需基因的定义[25], 必需基因不会出现组织特异性的表达.根据同源性预测方法(见1.3)得到了水稻的2 880个必需基因.根据定义, 预测的必需基因与组织特异基因之间的交集必定较小.将这些必需基因与识别的组织特异的基因进行比较, 发现识别的组织特异的基因与预测的必需基因只有极少数重叠.
2.3 水稻全基因组蛋白质相互作用 2.3.1 预测的水稻蛋白质相互作用网络水稻与6个参考物种的进化距离远近不一, 从各个参考物种中得到的直系同源基因的数量也有较大差异.图 5(a)展示了水稻与各个参考物种存在直系同源关系的基因的数目.图 5(b)是BLASTP程序发现的各个基因的直系同源蛋白质组的总数.共计得到6 560基因的116 393条相互作用关系.图 5(c)展示了这些蛋白质相互作用的来源分布.

|
图 5 同源映射方法预测的水稻蛋白质相互作用的分布 Figure 5 The distribution of predicted PPIs by Interolog method |
水稻与6个物种的进化距离由近到远分别是:拟南芥, 人类, 果蝇, 蠕虫, 酵母菌, 大肠杆菌.从图 5可看出, 水稻与拟南芥进化距离最近, 具有直系同源关系的基因数目最多, 在拟南芥中命中的直系同源基因占所有6个物种的33%, BLASTP检测到的直系同源物则占到46%.那么根据进化保守性原理, 水稻从这6个物种中继承来的蛋白质相互作用关系的数目应该由多到少, 即来自与拟南芥的最多, 来自于大肠杆菌的最少.从图 5(c)中可看出预测的蛋白质相互作用网络中来自拟南芥的相互作用最多.因此, 水稻的蛋白质相互作用网以及基因调控网络将会更类似于拟南芥.
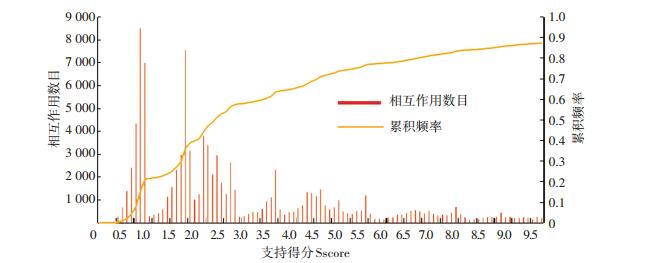
图 6展示了预测的蛋白质相互作用的支持得分Sscore的分布, 柱形图表示各个分数段中蛋白质相互作用的数目, 曲线图表示累积百分比.
|
图 6 支持得分Sscore的分布情况 Figure 6 The distribution of support score |
支持得分小于1表示源物种的直系同源物中没有找到最佳匹配的蛋白质对来支持目标物种中的蛋白质相互作用.根据支持得分含义以及分布情况, 选择Sscore>1的相互作用作为高可靠性的相互作用.经过筛选后, 预测的蛋白质相互作用总共包含6 088个基因, 98 696条相互作用关系.
对于上述筛选的蛋白质相互作用, 从网络的拓扑性质, GO注释, 共表达相关性3个方面进行可靠性分析与验证.
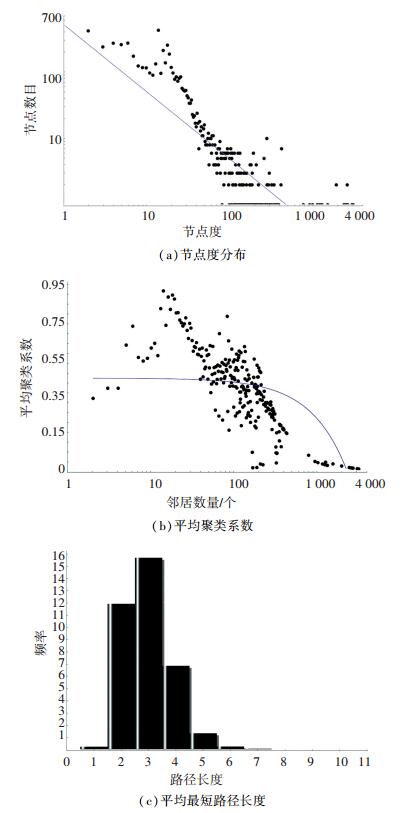
2.3.2 预测的网络的拓扑性质图 7是预测的蛋白质相互作用网络的具有代表性的网络拓扑结构的参数.
|
图 7 预测的蛋白质相互作用网络拓扑性质 Figure 7 Topological properties of predicted protein interaction network |
从网络中节点的度分布情况(图 7(a))可看出预测的网络呈现出无标度性质, 蛋白质相互作用网络的无标度特性使得生物对于环境变化或者刺激具有更强大的承受能力.从图 7(b)网络的平均聚类系数的分布情况可看出本文预测的网络的平均聚类系数较高.可见预测的网络具有与真实网络类似的模块化特性.图 7(c)是平均最短路径长度的分布情况, 预测的网络的最短路径长度多数落在2~5之间.由图 7(b)、(c)可知预测的网络是小世界网络.蛋白质相互作用网络的小世界特性有利于生物信号在网络中的迅速传播, 提高了生物响应外界刺激的速度与环境适应能力.从上述分析可看出预测的网络的拓扑性质与真实的蛋白质相互作用网络的拓扑性质类似.
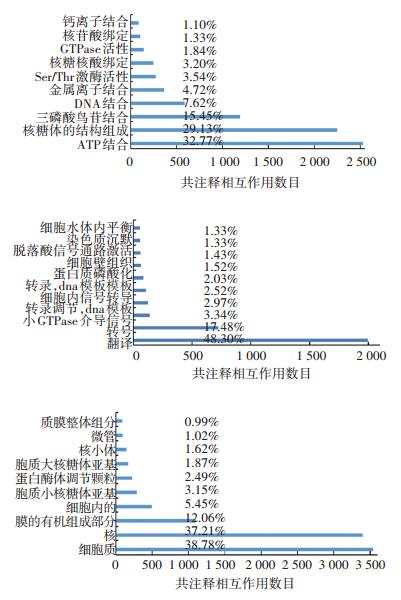
2.3.3 GO共注释分析图 8展示了具有相同注释的蛋白质相互作用的GO术语分布情况.GO术语分为分子功能(MF, Molecular Function), 生物过程(BP, Biological Process), 细胞组成(CC, Cellular Component)3个大类.具有相同注释的蛋白质发生相互作用的可能性更高.
|
图 8 共注释的蛋白质相互作用的GO术语分布 Figure 8 GO term distribution of co-annotation interactions |
目前水稻的GO注释的丰度较低, 在3个BP, CC, MF3个类别上的平均注释个数为1.44, 1.06, 1.60.对具有注释的蛋白质进行统计, 发现3个GO类别中具有相同注释的相互作用的比例分别为:CC中26.3%, BP中18.3%, MF中23.4%.从图 8(a)为的MF类别看, 超过60%的相互作用被标记为“绑定”功能.在BP类别(图 8(b))中发现中接近一半(48.3%)的蛋白质相互作用与翻译和转录相关.而在CC分类(图 8(c))中, 前三名分别被标记为细胞质(38.78%)与细胞核(37.21%), 膜结构(12.06%).这是蛋白质相互作用的主要场所.这说明预测蛋白质相互作用可靠性较高.
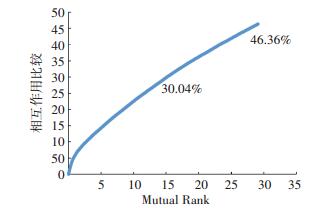
2.3.4 预测的蛋白质相互作用共表达情况表达水平的相关性一定程度上能够反应蛋白质相互作用的可靠程度.因此, 可以使用预测的蛋白质相互作用在转录层面的相关性来验证预测的蛋白质相互作用网络的可靠程度.ATTED-Ⅱ是一个针对拟南芥、水稻等模式植物的共表达信息库, 使用比相关系数更可靠的Mutual-Rank(MR, 相互排序)来衡量基因对之间的共表达关系.MR值越小, 说明基因之间的共表达关系越强.图 9是预测的蛋白质相互作用网络中所有蛋白质对对应的基因的共表达分布情况.从图上可看出30.04%的蛋白质对的共表达相关性排在前15%, 46.36%的蛋白质对的共表达相关性排在前30%.这说明预测的蛋白质相互作用有显著地共表达倾向, 即预测的蛋白质相互作用网络可信程度较高.
|
图 9 预测的网络中蛋白质对的共表达分布情况 Figure 9 Co-expression rank distribution of protein pairs in predicted network |
图 10展示了预测的结果PTSN4R与之前类似工作PRIN以及Matt预测的结果的异同.图 10(a)表示PTSN4R与Matt的工作以及PRIN中所涉及的基因的重叠程度.从图 10(a)可看出, PRIN以及Matt二者包含的基因重叠程度较高, 而本文预测的网络所涉及的基因与二者有较大不同.这是因为PTSN4R中有3 600个基因是先前的工作没有涉及的.图 10(b)展示了三项研究预测的蛋白质相互作用网络的重叠程度.从图 10(b)可看出, 本文预测的网络与前两项工作中预测网络重合程度也有较大差异, 新发现95 068条蛋白质相互作用关系, 极大的丰富了水稻相互作用研究的数据, 拓展了人们对水稻蛋白质相互作用的认识.经过分析发现, 新发现的相互作用主要来自拟南芥.这与近年来拟南芥蛋白质相互作用研究的快速发展有直接关系.随着蛋白质相互作用研究的不断发展, 同源映射方法还有较大的发展空间.
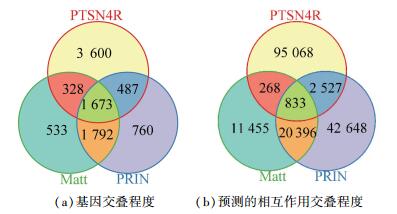
|
图 10 三项工作涉及的基因与预测相互作用重叠程度 Figure 10 Overlap of three research work |
为更好地对组织特异的蛋白质相互作用进行研究, 将PRIN、Matt等人预测的网络以及本文的预测结果合并构建了总的蛋白质相互作用网络.然后, 基于组织特异的基因和预测的蛋白质相互作用网络, 构建了水稻23个组织的蛋白质相互作用子网.最后, 根据共表达相关性筛选出了各个组织中可靠性较高的蛋白质相互作用.表 3列出了各个子网以及筛选的蛋白质相互作用的详细信息.
| 表 3 组织特异的蛋白质相互作用网络 Table 3 Tissue-specific protein interaction networks |
研究发现, 组织特异性的相互作用共计101 430条, 占全部预测的相互作用的58.6%.在这些组织特异的子网中, 花药、叶片、根以及胚乳组织的特异表达的基因由多个细胞类型合并而来, 因此形成的子网中相互作用数目最多.从表 3可看出各个子网都具有相对较高的聚类系数和网络中心度.即使是相互作用较少的细分组织(心皮, 外稃, 胚芽, 胚根, 小穗, 柱头)依旧具有较高的网络中心性.这说明各个组织特异的调控网络是构成整体小世界网络的一部分.
从表 3中可看到在各个组织特异的子网中发现的表达相关性较高的相互作用较少.进一步分析发现, 主要原因有3点:1)蛋白质相互作用网络覆盖度不高; 2)各个平台包含的基因集合不相同, 多平台数据整合过程中不交叉的基因被忽略; 3)整合的数据中存在较多检测失败的数据, 导致整体的表达相关性较低.
3 总结本文针对模式植物水稻, 提出了融合多组学数据构建组织特异性蛋白质相互作用网络的计算方法, 然后构建了23个水稻重要组织或者器官对应的组织特异的蛋白质相互作用子网(PTSN4R), 并从多个角度对预测结果进行分析, 说明了方法的合理性, 有效性.
组织特异的基因和蛋白质相互作用作为PTSN4R的核心, 拓展了人们对水稻组织特异基因和蛋白质相互作用网络的认知, 对基因调控机制和蛋白质功能的研究具有重要意义.我们期望PTSN4R能够促进组织特异的蛋白质相互作用的研究和对于水稻产量相关性状的调控机制的理解.
目前, 农作物相关的转录组以及蛋白质组学数据正在大量产出, 对于组织或者细胞特异表达的研究还在进一步的发展.后续可能的研究思路包括:将提出的方法应用于其他的动植物物种中; 研究更可靠的直系同源性度量方法, 提高准确性; 融合更丰富的数据提高相互作用网络的覆盖度及质量.
| [1] |
SHIMAMOTO K, KYOZUKA J. Rice as a model for comparative genomics of plants[J]. Annu Rev Plant Biol, 2002, 53(1): 399. DOI:10.1146/annurev.arplant.53.092401.134447 |
| [2] |
GOFF S A, RICKE D, LAN T H, et al. A draft sequence of the rice genome (Oryza sativa L.ssp.japonica)[J]. Science, 2002, 296(5565): 92. DOI:10.1126/science.1068275 |
| [3] |
SAKAI H, LEE S S, TANAKA T, et al. Rice Annotation Project Database (RAP-DB):an integrative and interactive database for rice genomics[J]. Plant & cell physiology, 2013, 54(2): e6. DOI:10.1093/pcp/pcs183 |
| [4] |
JIAO Yuling, TAUSTA S L, GANDOTRA N, et al. A transcriptome atlas of rice cell types uncovers cellular, functional and developmental hierarchies[J]. Nature genetics, 2009, 41(2): 258. DOI:10.1038/ng.282 |
| [5] |
FUJITA M, HORIUCHI Y, UEDA Y, et al. Rice expression atlas in reproductive development[J]. Plant & cell physiology, 2010, 51(12): 2060. DOI:10.1093/pcp/pcq165 |
| [6] |
WANG Lei, XIE Weibo, CHEN Ying, et al. A dynamic gene expression atlas covering the entire life cycle of rice[J]. The Plant Journal:For Cell and Molecular Biology, 2010, 61(5): 752. DOI:10.1111/j.1365-313X.2009.04100.x |
| [7] |
CHO K, SHIBATO J, KUBO A, et al. Genome-wide mapping of the ozone-responsive transcriptomes in rice panicle and seed tissues reveals novel insight into their regulatory events[J]. Biotechnology Letters, 2013, 35(4): 647. DOI:10.1007/s10529-012-1118-x |
| [8] |
HAMADA K, HONGO K, SUWABE K, et al. OryzaExpress:an integrated database of gene expression networks and omics annotations in rice[J]. Plant & Cell Physiology, 2011, 52(2): 220. DOI:10.1093/pcp/pcq195 |
| [9] |
KUDO T, AKIYAMA K, KOJIMA M, et al. UniVIO:a multiple omics database with hormonome and transcriptome data from rice[J]. Plant & Cell Physiology, 2013, 54(2): e9. DOI:10.1093/pcp/pct003 |
| [10] |
SATO Y, ANTONIO B, NAMIKI N, et al. Field transcriptome revealed critical developmental and physiological transitions involved in the expression of growth potential in japonica rice[J]. BMC Plant Biol, 2011, 11(10). DOI:10.1186/1471-2229-11-10 |
| [11] |
YAMAZAKI Y, SAKANIWA S, TSUCHIYA R, et al. Oryzabase:an integrated information resource for rice science[J]. Breeding Science, 2010, 60(5): 544. DOI:10.1270/jsbbs.60.544 |
| [12] |
NARSAI R, DEVENISH J, CASTLEDEN I, et al. Rice DB:an Oryza Information Portal linking annotation, subcellular location, function, expression, regulation, and evolutionary information for rice and Arabidopsis[J]. The Plant Journal:For Cell and Molecular Biology, 2013, 76(6): 1057. DOI:10.1111/tpj.12357 |
| [13] |
OBAYASHI T, OKAMURA Y, ITO S, et al. ATTED-Ⅱ in 2014:evaluation of gene coexpression in agriculturally important plants[J]. Plant & Cell Physiology, 2014, 55(1): e6. DOI:10.1093/pcp/pct178 |
| [14] |
LEE T, OH T, YANG S, et al. RiceNet v2:an improved network prioritization server for rice genes[J]. Nucleic Acids Research, 2015, 43(W1): W122-7. DOI:10.1093/nar/gkv253 |
| [15] |
CHANDRAN A K N, JUNG K H. Resources for systems biology in rice[J]. J Plant Biol, 2014, 57(2): 80. DOI:10.1007/s12374-014-0903-6 |
| [16] |
SMITH G R, STERNBERG M J. Prediction of protein-protein interactions by docking methods[J]. Curr Opin Struct Biol, 2002, 12(1): 28. DOI:10.1016/S0959-440X(02)00285-3 |
| [17] |
WANG Yongcui, WANG Jiguang, YANG Zhixia, et al. Sequence-based protein-protein interaction prediction via support vector machine[J]. J Syst Sci Complex, 2010, 23(5): 1012. DOI:10.1007/s11424-010-0214-z |
| [18] |
SHEN Juwen, ZHANG Jian, LUO Xiaomin, et al. Predicting protein-protein interactions based only on sequences information[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(11): 4337. DOI:10.1073/pnas.0607879104 |
| [19] |
MATTHEWS L R, VAGLIO P, REBOUL J, et al. Identification of potential interaction networks using sequence-based searches for conserved protein-protein interactions or "interologs"[J]. Genome Research, 2001, 11(12): 2120. DOI:10.1101/gr.205301 |
| [20] |
YU Haiyuan, LUSCOMBE N M, LU H X, et al. Annotation transfer between genomes:protein-protein interologs and protein-DNA regulogs[J]. Genome Research, 2004, 14(6): 1107. DOI:10.1101/gr.1774904 |
| [21] |
LO Yushu, CHEN Chunchen, HSU K C, et al.Rank-based interolog mapping for predicting proteinprotein interactions between genomes[C]// 2013 7th International Conference on Systems Biology (ISB).Huangshan, China: IEEE, 2013: 55.DOI: 10.1109/ISB.2013.6623794
|
| [22] |
GU Haibin, ZHU Pengcheng, JIAO Yinming, et al. PRIN:a predicted rice interactome network[J]. Bmc Bioinformatics, 2011, 12(161). DOI:10.1186/1471-2105-12-161 |
| [23] |
HO C L, WU Yinzhou, SHEN Hongbin, et al. A predicted protein interactome for rice[J]. Rice (N Y), 2012, 5(1): 15. DOI:10.1186/1939-8433-5-15 |
| [24] |
YANAI I, BENJAMIN H, SHMOISH M, et al. Genome-wide midrange transcription profiles reveal expression level relationships in human tissue specification[J]. Bioinformatics, 2005, 21(5): 650. DOI:10.1093/bioinformatics/bti042 |
| [25] |
ZHANG Ren, LIN Yan. DEG 5.0, a database of essential genes in both prokaryotes and eukaryotes[J]. Nucleic Acids Research, 2009, 37(Database issue): D455. DOI:10.1093/nar/gkn858 |
| [26] |
KRYUCHKOVA-MOSTACCI N, ROBINSON-RECHAVI M. A benchmark of gene expression tissue-specificity metrics[J]. Brief Bioinform, 2017, 18(2): 205. DOI:10.1093/bib/bbw008 |
 2018, Vol. 50
2018, Vol. 50


