2. 城市水资源与水环境国家重点实验室(哈尔滨工业大学),哈尔滨 150090
2. State Key Laboratory of Urban Water Resource and Environment(Harbin Institute of Technology), Harbin 150090, China
中国作为农业大国,农药的施用量逐年增加.近年来,因不合理使用及其难生化降解特性,农药常伴随着地表径流、干沉降和湿沉降等途径进入地表水和地下水,造成严重污染.阿特拉津(2-氯-4-乙胺基-6-异丙胺基-1,3,5-三嗪)是一种人工合成的化学除草剂,广泛应用于世界范围内,并严重危害自然界动植物的安全[1-4].在污水处理领域,如何高效降解阿特拉津是当前的热点问题.微生物修复技术因其成本低、不产生二次污染等特点已成为降解阿特拉津的重要技术之一.自20世纪80年代以来,至少存在19个属的微生物被证明具有降解阿特拉津的能力,包括节杆菌属、红球菌属、不动杆菌属、假单胞菌属、根瘤菌属、诺卡氏菌属等[5-9].然而,截至目前,能够高效降解阿特拉津的菌种资源非常有限.例如,Pooja Bhardwaja等[10]分离出的假单孢菌属EGD-AKN5能够将阿特拉津完全降解为二氧化碳,但其降解速率较低,难以进一步推广至实际应用.因此,为了将微生物修复技术应用于含有阿特拉津的废水处理中,有必要分离出具有高效降解能力的阿特拉津降解菌株,并对其降解特性加以研究.
本研究从长期受污染的吉林化工厂的土壤中分离得到一株高效的阿特拉津降解菌株,并对其进行鉴定.同时,研究了菌株在不同环境条件下的生长特性以及对阿特拉津的降解情况,并建立动力学模型用以表征菌株的生长和降解阿特拉津的能力,以期为微生物强化修复含有阿特拉津工农业废水的实际应用提供理论依据.
1 实验 1.1 细菌的分离与培养取吉林化工厂受污染土壤样品10 g加入至100 mL含有100 mg/L阿特拉津的无机盐培养基三角瓶中,在培养温度为30 ℃、pH7.0、摇床转速为150 r/min条件下摇床培养7 d,获得富集菌液.再取10 mL富集菌液加入至100 mL含有100 mg/L阿特拉津的无机盐培养基三角瓶中,重复此步骤3次.将富集后的菌液进行平板涂布,恒温培养箱培养3 d后获得单菌落.挑取如上单菌落进行平板三区划线,重复此步骤3次直至平板上出现形态大小相同的单菌落,其后将纯菌接入斜面培养基培养3 d,4 ℃保存.
其中,无机盐培养基为:0.9 g KH2PO4,6.5 g Na2HPO4·12H2O,3 g蔗糖,0.2 g MgSO4·7H2O,0.01 g FeSO4·7H2O,阿特拉津(固体阿特拉津溶解于甲醇溶液中制成阿特拉津甲醇储备液)以及1 mL微量元素液溶于1 000 mL蒸馏水中.无机盐固体培养基/斜面培养基是在无机盐的成分中另加入琼脂18 g/L.
微量元素液为:0.1 g CoCl2·6H2O,0.425 g MnCl2·4H2O,0.05 g ZnCl2,0.01 g NiCl2·6H2O,0.015 g CuSO4·5H2O,0.01 g Na2MoO4·2H2O,0.01 g Na2SeO4·2H2O溶于1 000 mL蒸馏水中.
1.2 菌株的鉴定将菌体于50 μL TaKaRa Lysis Buffer中离心后取上清作为模板,使用TaKaRa 16S rDNA试剂盒进行PCR扩增目的片段. PCR反应条件为:94 ℃预变性5 min,再经30个循环的94 ℃变性1 min,55 ℃退火1 min,72 ℃延伸1.5 min,而后再经过72 ℃延伸5 min.使用TaKaRa MiniBEST试剂盒切胶回收目的片段进行DNA测序.将测序结果采用Blast程序提交至GenBank与已知细菌序列进行比对,使用ClustalX及Mega软件进行系统发育树的绘制.最后将筛选菌株序列提交至NCBI数据库并保藏菌株.
对分离到的ZXY-1降解菌进行革兰氏染色,并根据伯杰氏细菌鉴定手册(第八版)进行常规生理生化实验[11].
1.3 菌株降解特性研究将菌体细胞接种至新鲜的液体培养基中(所用培养基为1.1所述培养基,培养至菌体浓度为108 CFU/mL),分别考察菌株在不同的温度(5、10、15、20、25、30、35、40 ℃)、pH(4.0、5.0、6.0、7.0、8.0、9.0、10.0)、摇床转速(0、50、100、150、200 r/min)以及接种量(1%、3%、5%、7%) (体积分数)环境下的生长特性以及降解特性,并确定最佳的生长及降解条件.单一变量改变的同时,固定温度25 ℃、pH 8.0、摇床转速150 r/min、接种量5%,初始阿特拉津质量浓度100 mg/L保持不变.9 h后取样,以不同温度、pH、摇床转速以及接种量为横坐标,以阿特拉津降解率及细菌OD600为纵坐标绘图分析.
1.4 动力学研究改变初始阿特拉津质量浓度(20、50、80、100、120、150、200、250、300、350、400、450、500 mg/L),在温度30 ℃、pH 8.0、摇床转速150 r/min、接种量3%条件下培养菌株ZXY-1.每隔1 h取样待测菌液的OD600值及阿特拉津质量浓度,研究菌株ZXY-1在不同初始质量浓度范围内的生长特性及降解阿特拉津特性.
在研究菌株ZXY-1降解时,常利用一级动力学方程进行数据拟合.即
| $ \rho {\rm{ = }}{\rho _0}{{\rm{e}}^{kt}}. $ | (1) |
对式两边同时取自然对数得
| $ \ln \rho = \ln {\rho _0} + kt. $ | (2) |
式中:k为一级动力学速率常数(h-1),ρ为t(h)时刻的质量浓度(mg/L),ρ0为初始质量浓度(mg/L),一级动力学半衰期为t1/2=ln2/k.
同时,利用零级动力学方程进行数据拟合,即
| $ \rho =-kt + {\rho _0}. $ | (3) |
式中:k为零级动力学速率常数(h-1),ρ为t(h)时刻的质量浓度(mg/L),ρ0为初始质量浓度(mg/L).
在好氧条件下,微生物生长动力学涉及底物的分解及微生物生物量的增长,采用式(4)计算菌株ZXY-1对数生长期的比生长速率(μ),并通过Haldane生长抑制模型(5)进行拟合,即
| $ \mu = \frac{1}{X}\frac{{{\rm{d}}X}}{{{\rm{d}}t}}. $ | (4) |
| $ \mu = \frac{{{\mu _{\max }}S}}{{{K_{\rm{s}}} + S + {S^2}/{K_{\rm{i}}}}}. $ | (5) |
式中:μ为比生长速率(h-1),X为细胞质量浓度(mg/L),μmax为最大比生长速率(h-1),S为底物质量浓度(mg/L),Ks为底物半饱和系数(mg/L),Ki为底物抑制系数(mg/L).
对式(5)求导,并令该方程的一阶导数为零,可得
| $ V = \frac{{{V_{\max }}}}{{1 + 2\sqrt {{K_{\rm{s}}}/{K_{\rm{i}}}} }}. $ | (6) |
此外,得到菌株达到最大比降解速率时对应的阿特拉津质量浓度, 即
| $ {S_{\max }} = \sqrt {{K_{\rm{s}}}{K_{\rm{i}}}} . $ | (7) |
阿特拉津的测定:以二氯甲烷作为萃取剂,通过液液萃取的方法萃取出样品中的阿特拉津,随后将样品进行10 000g离心10 min,再经0.22 μm滤膜过滤.利用高效液相色谱法测定阿特拉津的质量浓度,色谱条件为:C18柱,流动相为V(乙腈):V(水)=6:4,检测波长为220 nm,柱温30 ℃,流速1 mL/min,进样量20 μL.
菌体OD600值的测定:菌液OD600值为测定值与空白对照值的差值(以未接种菌液的选择性培养基OD600值作为空白对照值).同时,本实验将菌液离心后收集菌体,再与等量的无菌水混合后重悬菌体测定OD600值.
菌体细胞质量浓度的测定:将不同时期生长的菌液离心(10 000g, 5 min), 弃上清液获得菌体并干燥称重,建立细胞质量浓度X与OD600的线性关系:X=178.25×OD600 (mg/L).
2 结果与讨论 2.1 菌株ZXY-1的分离及鉴定从吉林化工厂受污染土壤样品中分离出多株阿特拉津降解菌,其中,菌株ZXY-1能够在11 h内完全降解初始质量浓度100 mg/L的阿特拉津,因此,将具有高效降解能力的菌株ZXY-1作为实验菌株.
菌株ZXY-1生理生化特性如表 1所示,其为好氧革兰氏阴性杆菌,结合氧化酶等生化实验,初步判定该菌株属于假单胞菌属.如图 1所示,基于16s rRNA序列及系统发育树可知,菌株ZXY-1与Pseudomonas sp.具有相似亲缘关系.因此,结合生理生化分析结果,菌株ZXY-1归为Pseudomonas.菌株Pseudomonas sp. ZXY-1已在GenBank中获得登录序列号为KT778823,且菌株已在“中国微生物菌种保藏管理委员会普通微生物中心”保藏,保藏编号为CGMCC No.10936.
| 表 1 菌株ZXY-1生理生化特性 Table 1 Physio-biochemical characteristics of strain ZXY-1 |

|
图 1 ZXY-1系统发育树 Figure 1 Pylogenetic tree of strain ZXY-1 |
如表 2所示,菌株ZXY-1在30 ℃、pH 7.0、摇床转速150 r/min、接种量5%的生长环境下降解效率为9.09 mg/(L·h).在相同的生长环境下,菌株ADP需要24 h才能完全降解初始质量浓度100 mg/L的阿特拉津,降解效率为4.17 mg/(L·h)[5].而菌株DAT1只能在48 h后实现初始质量浓度100 mg/L的阿特拉津的完全降解,降解效率仅为2.08 mg/(L·h)[12].由此可见,菌株ZXY-1具有高效降解阿特拉津的能力.
| 表 2 阿特拉津降解菌株降解效率比较 Table 2 Comparison of degradation efficiency of atrazine-degrading microorganisms |
将108 CFU/mL的菌液作为接种初始质量浓度,优化菌株ZXY-1的生长及降解条件,同时,研究菌株ZXY-1在不同温度、pH、摇床转速、接种量条件下的生长特性以及菌株对阿特拉津的降解特性.
温度是影响微生物新陈代谢最重要的因素,每种微生物都有其各自的最适生长温度范围,超过最适生长温度范围会使微生物的生长受到抑制作用[19].如图 2(a)所示,随着生长温度由5 ℃增加至30 ℃,菌株ZXY-1对阿特拉津的降解率由19.2%增加至100%,菌株的生长OD600值由0.11增加至0.72.再随着温度由30 ℃升高至40 ℃,阿特拉津降解率及生长OD600值均逐渐降低.因此,菌株ZXY-1最适生长温度为30 ℃.
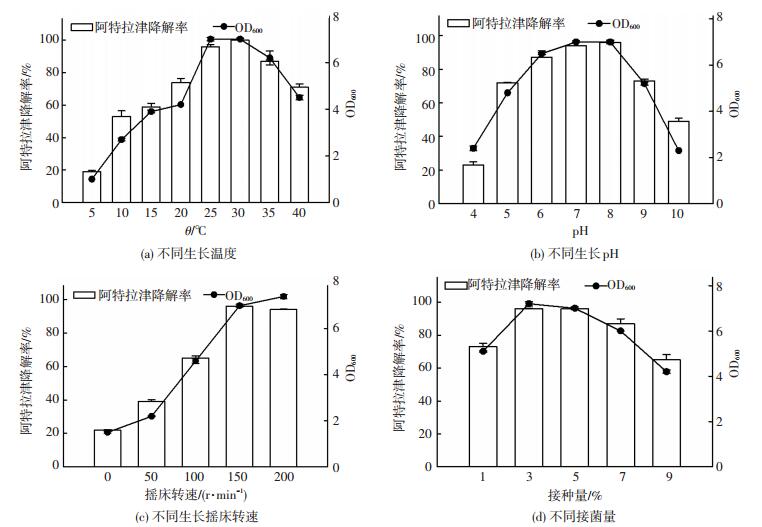
|
图 2 不同生长条件对ZXY-1生长及ZXY-1降解阿特拉津的影响 Figure 2 Different effects on the growth of strain ZXY-1 and atrazine degradation |
pH是影响微生物生长及物质降解的一个重要参数,过高或过低的pH都可能严重影响微生物对营养物质的运输和吸收[20].如图 2(b)所示,阿特拉津降解率及菌株生长OD600值随着pH的增加呈现先升高再降低的趋势.在初始pH为5~9内,菌株ZXY-1均能够在12 h内降解70%以上的阿特拉津,说明在偏酸性及偏碱性的环境条件下,菌株有较强的耐酸及耐碱能力.当初始pH为8.0时,阿特拉津的降解率为95.8%,OD600值为0.71,为最佳生长pH.即在pH 8.0的环境下既有利于菌株ZXY-1降解阿特拉津,又有利于菌株ZXY-1自身生长.
如图 2(c)所示,阿特拉津降解率及菌株生长OD600值随着摇床转速的增加而增加.当摇床转速达150 r/min时,阿特拉津降解率最高,可达96.4%.其原因是ZXY-1为好氧细菌,不同的摇床转速会导致培养基内不同的溶解氧含量,摇床转速过低会导致培养基内溶解氧含量较低,摇床转速越高,培养基内溶解氧含量越高,但转速过高会减弱酶与阿特拉津接触的机会,从而影响菌株的生长代谢.当摇床转速达150 r/min时,培养基内的溶解氧质量浓度基本达到饱和状态,此时为最佳的菌株生长溶解氧质量浓度,再进一步加快转速也难以提高溶解氧质量浓度.因此,150 r/min为最佳摇床转速.
如图 2(d)所示,阿特拉津降解率及菌株生长OD600值随着接种量的增加呈现先增大后降低的趋势.当接种量由1%增加至3%时,阿特拉津的降解及菌株的生长较为显著,其降解率及OD600值分别由73.2%增加至96.4%,0.51增加至0.72.然而,随着接种量增加至5%后,再继续增加接种量会导致阿特拉津降解率及细胞生长能力的降低.其原因是过量的菌体加入培养基后,菌体之间相互竞争有限的阿特拉津,从而导致菌体生长缓慢,生物量降低,菌体降解阿特拉津能力也随之减弱.因此,在接种量1%~9%范围内,3%为最适比例接种量.
综上,通过研究不同条件下菌株的生长以及底物的降解,得到最优的单因素条件为温度30 ℃、pH 8.0、摇床转速150 r/min、接种量3%.在此条件下,菌株ZXY-1能够在9 h内降解96.4%的阿特拉津,其降解效率为11.1 mg/(L·h).
2.3 不同初始质量浓度对菌株ZXY-1生长以及降解的影响不同的初始质量浓度会影响微生物的活性,高质量浓度的污染物可能会抑制微生物的生长,也有可能刺激微生物自身的繁殖[21].因此,在最优的单因素生长条件下,即温度30 ℃、pH 8.0、摇床转速150 r/min、接种量3%,考察菌株ZXY-1在不同阿特拉津初始质量浓度下对底物的降解能力以及菌株生长能力.
如图 3所示,在阿特拉津初始质量浓度为0~450 mg/L时,菌株能够在不同的时间内完全降解阿特拉津,但降解速率却各不相同.在阿特拉津质量浓度为20 mg/L时,5 h内阿特拉津能够完全被降解,并且没有延滞期的出现.当阿特拉津初始质量浓度为300 mg/L时,底物的降解受到明显的抑制,出现了5 h的延滞期,要达到完全降解需要延长降解时间至15 h.当阿特拉津初始质量浓度为500 mg/L时,降解延滞期约为10 h,且在25 h内不能将阿特拉津完全降解.因此,高质量浓度的阿特拉津会导致菌株降解阿特拉津延滞期的延长.
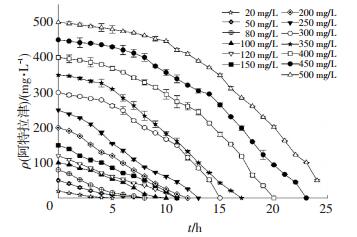
|
图 3 菌株在不同质量浓度下降解图 Figure 3 Time courses of the degradation of ZXY-1 at different initial atrazine concentrations |
如图 4所示,随着阿特拉津初始质量浓度的不断提高,菌体浓度也不断增加,由初始质量浓度20 mg/L时OD600值0.06增加至初始质量浓度500 mg/L时OD600值1.12.其原因是菌株随着时间通过利用阿特拉津自身繁殖,从而使生物量增加,并保持较高的生长代谢活性.菌株经过不同时间的延滞期后,能够较快地进入对数生长期.然而,细胞生长的延滞期随着初始阿特拉津质量浓度的增加而相应地延长.当初始阿特拉津为50 mg/L时,延滞期小于1 h,随着阿特拉津的质量浓度增加到400 mg/L,延滞期延长为5 h,这表明高质量浓度的阿特拉津会抑制菌体细胞的生长,导致菌株生长缓慢.
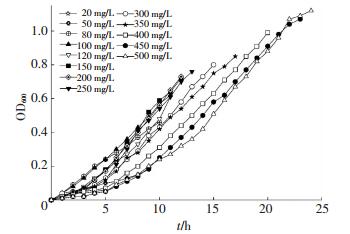
|
图 4 菌株在不同质量浓度下生长图 Figure 4 Time courses of the growth of ZXY-1 at different initial atrazine concentrations |
建立动力学模型对表征菌株的降解能力、理论研究和实际应用都有重要的指导意义.根据不同初始质量浓度下菌株ZXY-1的生长以及降解情况研究菌株降解阿特拉津的动力学性能.
如表 3所示,当阿特拉津初始质量浓度低于80 mg/L时,阿特拉津的生物降解过程符合一级动力学方程.此时,阿特拉津比降解速率与阿特拉津质量浓度呈正相关,阿特拉津的质量浓度成为影响菌株比降解速率的限制因子.在初始阿特拉津质量浓度为20、50、80 mg/L时,半衰期分别为1.12、1.54、1.58 h,降解速率常数分别为0.617、0.451、0.438 h-1.然而,Siripattanakul等[22]研究阿特拉津降解菌株MC1、MC2、MC3及J14a在初始质量浓度0.1 mg/L下的一级降解动力学,4株细菌的降解速率常数为0.11、0.10、0.06及0.08 h-1,其降解速率常数值远小于ZXY-1在低质量浓度下降解阿特拉津的速率常数.Marion Devers等[23]研究菌株Pseudomonas sp.ADP及菌株Chelatobacter heintzii在初始质量浓度15 mg/L下的一级动力学降解速率常数分别为0.052 7和0.021 9 h-1,而在初始质量浓度60 mg/L下的一级动力学降解速率常数分别为0.012 8和0.007 9 h-1,其数值也远小于ZXY-1.
| 表 3 菌株在阿特拉津初始质量浓度小于250 mg/L内的降解动力学方程 Table 3 Degradation kinetics equation of ZXY-1 at different initial atrazine concentrations (< 250 mg/L) |
另如表 3所示,当阿特拉津初始质量浓度在100~250 mg/L时,菌株的生物降解过程可用零级动力学模型进行拟合,此时的降解速率与底物质量浓度无关,阿特拉津的质量浓度已不是菌株降解的限制因子.在初始阿特拉津质量浓度为100、120、150、200、250 mg/L时,菌株降解半衰期分别为6.15、6.24、6.37、6.30、6.99 h.然而,刘娜等[24]研究了阿特拉津降解菌株W4在初始阿特拉津质量浓度为33、22和10 mg/L时,阿特拉津的降解符合零级降解动力学方程,其半衰期延长至2.2、2.0和2.3 d.当初始阿特拉津质量浓度高于250 mg/L时,R2也明显下降,菌株ZXY-1的降解基本不再符合零级动力学方程.
采用Haldane生长抑制模型模拟菌株在不同初始阿特拉津质量浓度下的生长动力学过程,通过Matlab 2015b软件利用非线性最小二乘法对实验数据进行拟合,拟合结果如图 5所示.菌株降解阿特拉津过程中的细胞生长动力学参数为:μmax=0.696 h-1,Ks=98.55 mg/L,Ki=142.6 mg/L.因此,菌株生长动力学方程为
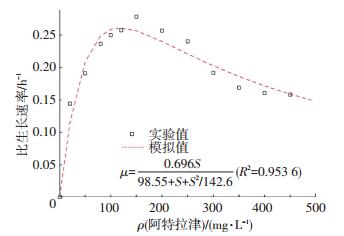
|
图 5 菌株在不同质量浓度下比生长速率 Figure 5 Specific growth rate of ZXY-1 at different atrazine concentrations |
| $ \mu = \frac{{0.696S}}{{98.55 + S + {S^2}/142.6}}. $ |
式中μmax为0.696 h-1,μmax值越高,菌株降解阿特拉津能力越强.Marion Devers等[25]研究了Pseudomonas sp. ADP在28 ℃、pH为6.6的生长环境下最大比生长速率为0.035 h-1,因此,可以进一步证明菌株ZXY-1的高效降解阿特拉津能力.
另根据式(7)可求得Sm=118.5 mg/L,即达到最大比降解速率时,最大底物质量浓度为118.5 mg/L,此质量浓度为菌株ZXY-1降解阿特拉津的最佳质量浓度.当阿特拉津质量浓度小于118 mg/L时,菌株对阿特拉津的比降解速率随着阿特拉津质量浓度的增加而升高;当阿特拉津质量浓度大于118 mg/L时,菌株对阿特拉津的比降解速率随着阿特拉津质量浓度的增加而降低,呈现底物抑制生长模式.若考虑实际应用,因实际工农业污水中的阿特拉津质量浓度为μg/L数量级,在水体环境中,阿特拉津的生物降解会按照一级反应动力学模式进行,较高质量浓度阿特拉津对菌株的生长和降解的影响可以忽略.因此,菌株ZXY-1能够为含有阿特拉津工农业废水的生物修复提供可行性选择.
3 结论1) 从吉林化工厂土壤样品中分离出一株高效阿特拉津降解菌ZXY-1,综合菌体形态特征、生理生化分析及16s rDNA分子鉴定结果,确定菌株属于Pseudomonas sp.
2) 菌株ZXY-1生长以及菌株ZXY-1降解阿特拉津的最优条件为温度30 ℃、pH 8.0、摇床转速150 r/min、接种量3%.在最佳条件下,菌株ZXY-1能够高效降解阿特拉津,其降解效率可达11.1 mg/(L·h).
3) 降解动力学分析表明在低质量浓度阿特拉津范围内(< 80 mg/L),降解过程符合一级降解动力学模型;在阿特拉津质量浓度为100~250 mg/L内,降解过程符合零级降解动力学模型.过高的阿特拉津质量浓度能够抑制菌株ZXY-1的细胞生长,Haldane生长抑制模型能够较好地拟合菌株以阿特拉津为单一底物的生长动力学性能,其相关动力学参数为μmax=0.696 h-1,Ks=98.55 mg/L,Ki=142.6 mg/L(R2=0.953 6).
| [1] |
SU Junfeng, ZHENG Shengchen, HUANG Tinglin, et al. Characterization of the anaerobic denitrification bacterium Acinetobacter sp. SZ28 and its application for groundwater treatment[J].
Bioresour Technol, 2015, 192: 654-659.
DOI: 10.1016/j.biortech.2015.06.020 |
| [2] |
CAI B, HAN Y, LIU B, et al. Isolation and characterization of an atrazine-degrading bacterium from industrial wastewater in China[J].
Lett Appl Microbiol, 2003, 36(5): 272-276.
DOI: 10.1046/j.1472-765X.2003.01307.x |
| [3] |
VANRAES P, WILLEMS G, NIKIFOROV A, et al. Removal of atrazine in water by combination of activated carbon and dielectric barrier discharge[J].
J Hazard Mater, 2015, 299: 647-655.
DOI: 10.1016/j.jhazmat.2015.07.075 |
| [4] |
郭火生, 王志刚, 孟冬芳, 等. 阿特拉津降解菌株DNS32的降解特性及分类鉴定与降解途径研究[J].
微生物学通报, 2012, 39(9): 1234-1241.
GUO Huosheng, WANG Zhigang, MENG Dongfang, et al. Degradation characteristics and identification and the degradation pathway of the atrazine-degrading strain DNS32[J]. Microbiology China, 2012, 39(9): 1234-1241. |
| [5] |
MANDELBAUM R T, ALLAN D L, WACKETT L P. Isolation and characterization of a Pseudomonas sp. that mineralizes the s-triazine herbicide atrazine[J].
Appl Environ Microbiol, 1995, 61(4): 1451-1457.
|
| [6] |
SINGH P, SURI C R, CAMEOTRA S S. Isolation of a member of Acinetobacter species involved in atrazine degradation[J].
Biochem Biophys Res Commun, 2004, 317(3): 697-702.
DOI: 10.1016/j.bbrc.2004.03.112 |
| [7] |
FAZLURRAHMA N, BATRA M, PANDEY J, et al. Isolation and characterization of an atrazine-degrading Rhodococcus sp. strain MB-P1 from contaminated soil[J].
Lett Appl Microbiol, 2009, 49(6): 721-729.
DOI: 10.1111/lam.2009.49.issue-6 |
| [8] |
El S T, MARION D L, FREDERIQUE C, et al. Evidence of atrazine mineralization in a soil from the Nile Delta: Isolation of Arthrobacter sp. TES6, an atrazine-degrading strain[J].
International Biodeterioration & Biodegradation, 2011, 65(8): 1249-1255.
|
| [9] |
WANG Jinhua, ZHU Lusheng, WANG Qi, et al. Isolation and characterization of atrazine mineralizing Bacillus subtilis strain HB-6[J].
PLoS One, 2014, 9(9): e107270.
DOI: 10.1371/journal.pone.0107270 |
| [10] |
BHARDWAJ P, SHARMA A, SAGARKAR S, et al. Mapping atrazine and phenol degradation genes in Pseudomonas sp EGD-AKN5[J].
Biochem Eng J, 2015, 102: 125-134.
DOI: 10.1016/j.bej.2015.02.029 |
| [11] |
DONG X, CAI M.
Identification manual of common bacteria system[M]. Beijing: Science Press, 2001.
|
| [12] |
WANG Qingfeng, XIE Shuguang. Isolation and characterization of a high-efficiency soil atrazine-degrading Arthrobacter sp. strain[J].
International Biodeterioration & Biodegradation, 2012, 71: 61-65.
|
| [13] |
NAGY I, COMPERNOLLE F, GHYS K, et al. A single cytochrome P-450 system is involved in degradation of the herbicides EPTC (S-ethyl dipropylthiocarbamate) and atrazine by Rhodococcus sp. strain NI86/21[J].
Appl Environ Microbiol, 1995, 61(5): 2056-2060.
|
| [14] |
STRUTHERS J K, JAYACHANDRAN K, MOORMAN T B. Biodegradation of atrazine by Agrobacterium radiobacter J14a and use of this strain in bioremediation of contaminated soil[J].
Appl Environ Microbiol, 1998, 64(9): 3368-3375.
|
| [15] |
LI Qingyan, LI Ying, ZHU Xikun, et al. Isolation and characterization of atrazine-degrading Arthrobacter sp. AD26 and use of this strain in bioremediation of contaminated soil[J].
J Environ Sci (China), 2008, 20(10): 1226-1230.
DOI: 10.1016/S1001-0742(08)62213-5 |
| [16] |
WANG Jinhua, ZHU Lusheng, LIU Aiju, et al. Isolation and characterization of an Arthrobacter sp. strain HB-5 that transforms atrazine[J].
Environ Geochem Health, 2011, 33(3): 259-266.
DOI: 10.1007/s10653-010-9337-3 |
| [17] |
代先祝, 胡江, 蒋建东, 等. 污染土壤中原位阿特拉津降解菌的分离和鉴定[J].
土壤学报, 2006, 43(3): 467-472.
DAI Xianzhu, HU Jiang, JIANG Jiandong, et al. Isolation and identification of in situ atrazine-degrading bacteria from contaminated soils[J]. Acta Pedologica Sinaca, 2006, 43(3): 467-472. DOI: 10.11766/trxb200503160316 |
| [18] |
ZHANG Ying, JIANG Zhao, CAO Bo, et al. Metabolic ability and gene characteristics of Arthrobacter sp. strain DNS10, the sole atrazine-degrading strain in a consortium isolated from black soil[J].
International Biodeterioration & Biodegradation, 2011, 65(8): 1140-1144.
|
| [19] |
陈佳亮, 刘晓文, 张雅静, 等. 耐性细菌的分离鉴定及重金属污染修复初步研究[J].
生态环境学报, 2014, 23(7): 1199-1204.
CHEN Jialiang, LIU Xiaowen, ZHANG Yajing, et al. Isolation and identification of four tolerance bacteria and the preliminary study for pollution remediation of heavy metals[J]. Ecology and Environmental Sciences, 2014, 23(7): 1199-1204. |
| [20] |
郑菲菲, 胡江, 辛华东, 等. N-乙烯吡咯烷酮降解菌的筛选降解及其竹炭固定化[J].
化境工程学报, 2016, 10(10): 5593-5599.
ZHENG Feifei, HU Jiang, XIN Huadong, et al. Isolation and degradation characteristics of a N-vinylpyrrolidone degrading strain ZF1 and its immobilization on bamboo-based carbon[J]. Techniques and Equipment for Environmental Pollution Control, 2016, 10(10): 5593-5599. |
| [21] |
辛蕴甜. 石油降解菌的降解性能, 固定化及降解动力学研究[D]. 上海: 东华大学, 2013.
XIN Yuntian. Study on degradation property, immobilization and degradation kinetics of oil degrading bacteria[D]. Shanghai: Donghua University, 2013. |
| [22] |
SIRIPATTANAKUL S, WIROJANAGUD W, MCEVOY J, et al. Atrazine degradation by stable mixed cultures enriched from agricultural soil and their characterization[J].
J Appl Microbiol, 2009, 106(3): 986-992.
DOI: 10.1111/jam.2009.106.issue-3 |
| [23] |
DEVERS M, SOULAS G, FABRICE M L. Real-time reverse transcription PCR analysis of expression of atrazine catabolism genes in two bacterial strains isolated from soil[J].
J Microbiol Methods, 2004, 56(1): 3-15.
DOI: 10.1016/j.mimet.2003.08.015 |
| [24] |
刘娜, 毕海涛, 任何军, 等. 阿特拉津低温降解菌的筛选及降解机理研究[J].
吉林大学学报(地球科学版), 2009, 39(5): 893-898.
LIU Na, BI Haitao, REN Hejun, et al. Isolation of atrazine degrading low temperature bacteria and researching of atrazine biodegradation[J]. Journal of Jilin University, 2009, 39(5): 893-898. |
| [25] |
DEVERS M, ROUARD N, FABRICE M L. Fitness drift of an atrazine-degrading population under atrazine selection pressure[J].
Environ Microbiol, 2008, 10(3): 676-684.
DOI: 10.1111/emi.2008.10.issue-3 |
 2018, Vol. 50
2018, Vol. 50


