2. 城市水资源与水环境国家重点实验室(哈尔滨工业大学), 哈尔滨 150090
2. State Key Laboratory of Urban Water Resources and Environment (Harbin Institute of Technology), Harbin 150090, China
微生物广泛分布于自然和人工环境中,构成了复杂多样的微生物生态系统,在微生物种群间、微生物与环境间存在着多种互作关系,在调节生态系统和物质循环过程中发挥关键作用[1].微生物通过不同的代谢途径实现水中污染物转化和降解,从而实现了污水生物处理和水体自然修复过程.污水处理过程包括多样性的微生物种群,如发酵细菌、硝化菌、反硝化菌、厌氧氨氧化菌、聚磷菌、电化学活性菌和产甲烷菌等[2-5],微生物群落结构直接影响污染物生物转化速度和末端产物.生态因子和生物反应器运行条件会影响微生物的活性和生理生态功能,从而影响生物反应器的运行效果[6-9].因此,全面了解自然环境和生物处理系统中的微生物群落结构和功能及其种群互作机制,可为有效对污水生物处理过程进行定向调控提供微生物学依据.
据评估,自然界有99%的微生物未获得纯培养[10],这些未培养的微生物被称为“微生物暗物质”(microbial dark matter)[11].基于核酸测序的分子生物学技术为研究未培养微生物的生理生态特性和代谢途径提供了思路和方法[12].近年来兴起的基于高通量核酸测序技术的宏基因组学、宏转录组学和单细胞基因组学,为不同环境中未培养微生物的特性和代谢功能研究提供了有效的工具[13-14].1998年,Handelsman等[15]首次提出了宏基因组学(metagenomics)的概念,宏基因组学是指自然或人工环境样品的微生物群体基因组,通过核酸测序分析进行功能基因筛选,探究微生物种群多样性、物种进化、代谢功能、种群互作以及与环境互作的研究方法[16].宏转录组学(metatranscriptomics)是从RNA水平研究微生物群落在某一功能状态下的全部转录本的种类、功能和表达活性,能够直接反映微生物群落的代谢活性,为鉴定和分析微生物群落中的关键功能基因和代谢途径提供了新方法[17-18].近十年来,关于宏基因组学和宏转录组学的研究论文大幅度增长(图 1).利用宏基因组学和宏转录组学技术为获取未培养微生物基因及其可能的代谢途径提供了可能.
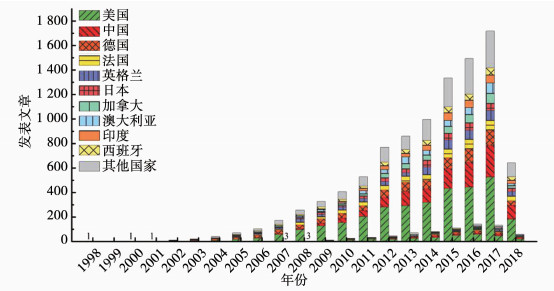
|
宏基因组(左侧)和宏转录组(右侧)发表文章数,检索关键词为metagenome和metagenomic;年份右侧对应宏转录组发表文章数,检索关键词为metatranscriptome和metatranscriptomic,统计来源于web of science核心合集 图 1 宏基因组学和宏转录组学近20年发表文章统计 Fig. 1 Publication statistics of metagenomics and metatranscriptomics in recent twenty years |
随着高通量核酸测序技术和生物信息学的发展,宏组学(meta-omics)技术已经成为污水生物处理系统微生物组研究的热点之一,为研究微生物群落结构和功能,揭示微生物群落对生态环境的响应和调控机制提供了重要工具[19].本文回顾了核酸测序技术的发展,综述了近年来基于高通量核酸测序技术的宏基因组学和宏转录组学方法在生物脱氮、强化生物除磷及微生物电化学系统中的应用,展望了宏组学技术在污水生物处理系统研究的应用前景.
1 核酸测序技术的发展 1.1 Sanger核酸测序测序技术在环境微生物研究中发挥着重要作用,测序技术不断取得革命性的突破促使大数据获取成本显著降低,极大地推进了相关研究的发展.第一代DNA测序技术是由Sanger发明的经典双脱氧核苷酸末端终止测序法[20].通过Sanger测序能获取较长读长的序列(500~1 000 bp),原始数据准确率高达99.99%,一直被作为测序质量的参考标准.Sanger测序是1975—2005年被广泛应用的测序技术,基于Sanger测序的第一个细菌基因组测序于1995完成[21].在人类基因组计划中,第一代测序技术发挥了重要作用[22].较低的测序通量和较慢的测序速度限制了Sanger测序法在基因组学研究中的应用,该技术在测序量需求不高时仍然可被使用.
1.2 高通量核酸测序技术2005年,基于焦磷酸测序原理的454测序平台的出现开启了二代测序(next generation sequencing,NGS)的时代[23].NGS的主要步骤包括DNA的提取;测序文库的体外构建;实现DNA片段的体外扩增;自动化测序.这种高通量技术不需要繁琐的克隆过程,可以检测几十万到几百万条短读长序列,使测序成本显著降低,测序效率大幅提高.随着高通量测序技术进入市场,基于在基质上固定DNA的不同方法,逐步形成了以Roche 454焦磷酸测序、Solid测序和Illumina测序为代表的商业化测序平台.Roche 454平台采用焦磷酸测序原理[23],Roche 454系统拥有长读长的优点和相对快的速度,与后来出现的二代测序技术相比其具有通量低和测序成本较高的不足.基于双碱基编码系统的Solid测序平台通量较高但读长短,采用的是连接测序的方式[24].Illumina测序平台采用的是边合成边测序的策略[25],测序具有高质量的数据、较低的样品需求以及简单的操作流程等优点.
1.3 单分子核酸测序技术二代核酸测序技术的发展对微生物生态学和组学研究起到了重大推动作用,使得土壤、海洋、污水等环境样品被大量测序和分析,促进了对环境微生物及其功能多样性的了解[26].然而,PCR扩增的偏好性往往会导致扩增后与扩增前DNA片段的相对比例存在偏差.此外,NGS测序的读长较短,对于高GC含量基因组测序、序列拼接、基因组组装等都具有很多挑战.为了弥补NGS技术中存在的缺陷,三代测序技术直接针对单个DNA分子测序而不需要进行PCR扩增,在高GC含量基因组测序方面也具备优势.
较早商业化测序平台是PacBio公司的单分子实时测序(single molecule real-time,SMRT)技术[27].由Oxford Nanopore Technology推出的纳米孔测序仪MinION是第一个运用纳米孔技术的商业化平台,通过测量电导率的变化来确定DNA序列[28].其测序读长长,可实时分析通过测序仪的片段,由于不需要对碱基进行荧光修饰,在一定程度上降低了成本,而采用电生理学的检测机制可以减少由酶活引起的偏差.尽管单分子测序技术仍在迅速发展,相对成熟的第二代测序技术由于价格优势仍被大量应用,目前仍处于多种测序平台共存的时期.
2 宏组学技术在污水生物处理过程微生物研究中的应用生物污水处理技术主要利用微生物的物质代谢,实现碳、氮、磷、硫等营养循环和生物转化[29].通过基因组测序可以获取纯菌种的功能基因和代谢特征信息,由于仅有1%的微生物可被分离[10],无法全面获取整个群落的功能信息,基于纯菌种解析微生物种群互作仍然非常困难,如何将微生物群落作为整体来探究其功能仍然是一大挑战.随着测序技术的进步和测序成本的降低,从分子水平上探究复杂微生物群落及钓取未培养微生物的基因组成为可能.宏组学分析主要包括样品收集和核酸提取、建库及高通量测序、数据预处理、序列组装及基因预测、物种及功能注释、多样品统计分析和实验验证等过程(图 2).将环境中获得的宏基因组序列进行聚类(genome-binning)可以重建单个微生物的基因组[30],从而揭示未培养微生物的生态适应机制和新陈代谢功能等.基于宏转录组学研究可分析微生物群落的基因表达,阐明不同生态条件下基因表达调控机制[31].

|
RPKM(reads per kilobase per million mapped read); FPKM (fragments per kilobase of transcript sequence per millions base pairs sequenced); KEGG (kyoto encyclopedia of genes and genomes); eggNOG (evolutionary genealogy of genes: Non-supervised orthologous groups) 图 2 环境样品的宏组学技术分析流程 Fig. 2 Process of meta-omic analysis for environmental samples |
宏组学(meta-omics)技术为微生物群落功能研究提供了新的方法.这些技术被用于揭示活性污泥和微生物生物膜群落中优势功能种群的生理特性,发现潜在功能基因,以及功能基因表达的调控等[32-33].宏组学技术已经被大量应用于污水生物处理技术的微生物群落结构与功能研究(表 1).
| 表 1 宏组学在污水生物处理系统微生物群落研究中的应用 Tab. 1 Applications of meta-omics in investigation on microbial community of biological wastewater treatment systems |
生物脱氮工艺是污水处理技术中重要的环节之一,传统生物脱氮主要通过硝化和反硝化作用来实现,多样性的微生物种群参与氮转化过程[34].通过宏基因组学分析,可以研究污水生物处理工艺中未培养脱氮微生物及其氮代谢相关功能基因[2, 35-36].宏基因组学分析为探索活性污泥等复杂的生态系统提供了有力的支持[36],两个不同规模脱氮系统的氨氧化细菌含量均比氨氧化古菌丰富,代谢途径总体相似,而在某些通路特异基因的数量上有所不同.在制革污水处理厂中发现好氧和厌氧污泥中的反硝化菌主要包括Thauera, Paracoccus, Hyphomicrobium, Comamonas和Azoarcus,宏基因组学分析证实氮代谢相关功能基因amoA,nirK,nirS和nosZ在好氧污泥中的丰度高于厌氧污泥[2].外源因子如重金属和高浓度盐会对脱氮系统产生影响[37-38].铬会降低细菌群落生物多样性和潜在的反硝化菌的种类,虽然铬对细菌群落的功能影响不明显,但改变了特定反硝化基因的丰度,随着铬浓度的增加nirS的丰度提高而nirK和norB的丰度降低[37].盐胁迫会降低反硝化菌的生物多样性,无盐胁迫时以硝酸还原酶基因为主的反硝化基因丰度增加,而nirS基因在亚硝酸盐还原中比nirK发挥更重要的作用[38].
厌氧氨氧化(anaerobic ammonium oxidation, Anammox)作为一种节能的污水脱氮技术受到广泛关注,而了解厌氧氨氧化菌的进化关系、生理特性及代谢途径对于Anammox技术应用非常关键[3].由于无法获得纯培养Anammox菌,宏基因组学技术成为研究Anammox菌功能基因和代谢特征的有效手段[39].了解反应器启动阶段Anammox菌群的群体感应有利于强化反应器的快速启动[40],在启动初期细菌交流比高负荷阶段更活跃,这与细菌多样性的逐渐减少相关.调控种内信号分子AHL (acyl homoserine lactone)产生的基因hdtS以及调控种内和种间信号分子DSF (diffusible signal factor)产生的基因rpfF是Anammox菌群内的关键交流基因(bacterial communication gene),高丰度的hdtS和rpfF推动了Anammox菌群内的信息交流,而且富含交流基因的细菌更倾向与相似功能的细菌产生相互作用[40].宏基因组聚类获得了来自大型Anammox反应器样本的23个基因组草图为全面系统认识功能微生物群落提供有效方法[41].通过比较不同Anammox培养物的系统发育和功能特点,发现生物膜和颗粒污泥中Anammox菌群的丰度和多样性要高于絮状污泥,但Candidatus Kuenenia均为其中的优势菌[42].同时,在生物膜和颗粒污泥中高效鉴定了代谢相关的基因,其中hzs基因在生物膜中的丰度要显著高于颗粒污泥.基于两个隶属于Candidatus Brocadia的新种的高质量基因组,重建了其Anammox分解代谢途径,对代谢通路的分析表明两个Anammox菌适合在无机氮环境中生长[43].宏基因组测序表明两个Anammox菌在悬浮和附着生长的反应器内都得到有效富集,很大程度上是由于接种物均直接或间接来自于富含Ca. Brocadia fulgida的反应器,这也证明了接种物对于Anammox菌群富集具有重要影响[8].
基因丰度与基因表达水平之间可能存在差异,因此,需要结合宏基因组学和宏转录组学分析活性污泥样本的微生物群落结构及功能基因的表达情况[31].在与氮代谢紧密相关的硝化、反硝化、氨化和固氮过程中,反硝化相关基因的丰度和基因表达活性均占主导地位,尽管硝化相关基因在宏基因组数据中丰度较低,但宏转录组数据显示群落表达了相对高水平的硝化活性,特别是氨单加氧酶和羟胺氧化酶具有很高的cDNA/DNA比例,这突出了基因表达分析在检测群落内活跃物种方面的重要性[31].在Genome binning获得17个厌氧氨氧化菌和异养菌的基因组草图基础上,结合功能菌群的代谢重建及相关基因的表达可揭示其潜在的生态作用和相互作用[44].含有抗生素的污水处理也是研究的热点,抗生素会影响脱氮微生物功能基因的相对表达水平,造成氨氧化相关的amoA和hao基因表达量显著降低,而nir和nos等基因代表的反硝化活性显著增加[45].目前,多组学分析已经成为生物脱氮研究领域的重要手段之一,随着研究的深入得到更广泛的应用.
2.2 利用宏组学技术揭示强化生物除磷种群关系目前,污水处理的趋势是同步实现资源回收,而强化生物除磷(enhanced biological phosphorus removal, EBPR)因经济、有效的优势可用于磷的去除和回收[46].实现EBPR高效稳定运行需要深入了解系统内的主要功能微生物及其代谢机理,许多基于组学的研究取得了一些突破性的进展[47-48].Candidatus Accumulibacter phosphatis[49]是目前污水生物处理领域重要且研究较多的聚磷菌(polyphosphate accumulating organisms,PAOs),基于宏基因组测序从两个富含Ca. Accumulibacter phosphatis的污泥样品中获得了该微生物近乎完整的基因组,检测到的高亲和性磷酸盐转运蛋白基因有利于磷的高效去除[47].通过综合的代谢重建,针对EBPR中的一些争议问题提出了见解,如一种新的细胞色素b/b6可以再氧化由琥珀酸脱氢酶产生的还原醌,从而促使厌氧条件下通过TCA循环产生电子传递链上的还原力,这为后续的组学研究奠定了基础[47].基于ppk1 基因的进化分析可将Ca. Accumulibacter phosphatis划分为TypeⅠ和TypeⅡ两个进化分支[48],其中每个分支又包含几种子分枝[4],而且相对于地理位置,环境条件对聚磷菌种群多样性分布起着关键作用[9].通过宏基因组构建基因组草图可以比较不同环境下的聚磷菌的基因组成,在Ca. Accumulibacter两种代表性分支菌株中EBPR代谢相关的功能基因没有明显差异,但是两个基因组整体之间缺乏明显的共线性[50].
聚糖菌(glycogen accumulating organisms,GAOs)与PAOs具有类似的代谢特征,由于不具备除磷能力,在与PAOs竞争底物过程中容易导致系统除磷效率降低[51].目前,研究广泛的聚糖菌为Candidatus Competibacter,从两个实验室规模的反应器中构建的Ca.Competibacter denitrificans和Candidatus Contendobacter odensis基因组分别属于它的分支1和5[52].两个基因组之间都含有挥发性脂肪酸代谢相关的基因,但在糖代谢和氮代谢方面存在一些差异.同PAOs相比,GAOs在磷代谢途径上缺乏Pit磷酸盐转运蛋白,这也可能是导致两者生理代谢差异的原因之一[52].一个新的聚糖菌Propionivibrio被富集在大型EBPR污水处理系统中[53].PAOs和GAOs通常在系统发育上是不相关的,而基因组注释表明Propionivibrio与PAOs密切相关且同在反应器内处于优势种群地位,这将为研究EBPR微生物群落以及PAO和GAO的种群关系提供重要意义[53].
通过对生物反应器主要种群及其功能基因的研究,了解群落结构和潜在代谢途径将有助于提高EBPR的运行效果.磷酸盐代谢和生物膜形成相关基因的富集反映了EBPR工艺具有定向富集的作用[54].通常EBPR需要通过厌氧/好氧或厌氧/缺氧阶段交替运行,而在一个污泥处理系统中,非反硝化聚磷菌在非厌氧条件下实现了磷的有效去除,这对于改进系统设计提供了新思路[55].
不同聚磷菌Ca.Accumulibacter phosphatis比较基因组研究,揭示了一些必需的核心代谢基因是保守的.宏转录组分析揭示了EBPR在不同生态条件下相关功能基因的表达水平差异[56].通过比较CAP IB HKU-1在厌氧阶段与好氧阶段差异表达的基因,发现同化型硫酸盐还原和磷吸收相关基因显著上调表达,而N2O、PHA合成和乙酰辅酶A相关基因显著下调表达[56].共表达基因集的鉴定表明在EBPR循环过程中,转录调控直接影响了PAO好氧与厌氧代谢[57].温度被视为影响PAO和GAO种间竞争的一个重要因素[58],通过宏组学分析,在一个热带气候下具有EBPR活性的系统中却发现高丰度的PAO和GAO共存,并且TCA循环和糖酵解途径相关的基因高效表达,这凸显了组学对于生态系统中种群竞争动态变化研究的重要性[59].随着EBPR系统内许多功能菌基因组的获得,未来通过宏组学技术的联合分析可以更加深入探究微生物在不同条件下的代谢机制.
2.3 基于宏组学解析电化学活性微生物生物膜生理与生态微生物电化学系统(microbial electrochemical system,MES)作为新兴的污水处理技术得到了迅速发展,它能直接从生物质或有机废物回收清洁的电能,具有集污水净化和产能一体的优点[5].其接种物大多数来源于生活污水、厌氧污泥和水体沉积物等,在各类环境因子影响下,系统内的微生物种群丰富多样[6, 7, 60-61].宏基因组学已经成为探究MES中微生物种群动态和代谢潜能的有力工具[62-63].MES中发挥电催化活性的主要是富集在阳极的电活性菌(electroactive bacteria, EAB).
结合16S rRNA基因和宏基因组序测序可以探寻关键功能种群[64],Geobacter psychrophilus适合在根际沉积型MFC(microbial fuel cell)的阳极上生长,并通过与其他微生物的互养作用实现产电.以甲醇为底物的MFC微生物群落中,Dysgonomonas,Sporomusa和Desulfovibrio大量出现在阳极和阴极生物膜及悬浮液中,而Geobacter仅存在于阳极生物膜[65].基于细菌的生理特性及宏基因组重建的相关代谢途径,推断Sporomusa和Geobacter互作菌群的厌氧分解促进了甲醇转化为电能,其中Sporomusa将甲醇转化为乙酸,随后被Geobacter利用来产电[65].
基因组聚类分析(genome-binning)也应用于重建MES阳极和阴极核心微生物的基因组[32, 66].从MFC的阳极生物膜群落中获取4种高丰度微生物的基因组,其中两组属于产电微生物Desulfobulbaceae[32].从微生物电合成系统中聚类出Sulfurospirillum sp属菌株的基因组草图,其中含有参与氧化亚氮还原的nosZDFLY基因,表明该菌株具有潜在的反硝化作用[67].类似的聚类方法也用于表征阴极相关的微生物群落[68],并重建了生物阴极的核心微生物Candidatus Tenderia electrophaga的基因组,推测其可以利用来自阴极的电子固定CO2[69].特别地,其基因组的相对丰度与电流增加呈现正相关性,进一步表明该菌可能为电能自养菌[70].而Marshall等[66]从利用二氧化碳产氢产乙酸的微生物电合成系统中组装出13个基因组,并构建了群落中优势菌的代谢模型,发现Acetobacterium是具有固碳功能的关键菌.
此外,相关研究已经利用宏转录组的方法来分析MES微生物的基因表达情况,从而加深对群落功能的认知.两个由宏基因组聚类获得的与EABs相关的物种Desulfobulbaceae和Desulfuromonadales在对不同电化学应激时表现出显著的基因表达活性,其中细胞色素C相关基因在提高产电过程中显著上调表达[32].许多未知功能的基因同样在刺激下上调表达,表明阳极微生物有可能利用其他未知成分参与了胞外电子传递[32].另一方面,通过重建阳极微生物菌群间的代谢网络,可评价不同功能微生物间的互作关系[71].稳定运行下,Desulfobacteraceae和Methanosarcinaceae的异化硫酸盐还原和甲烷合成基因显著高效表达.然而,这些基因的表达水平在改变电子传递速率时没有显著变化,表明这些微生物不参与产电,可能同EAB存在竞争关系[71].
生物阴极中的某些微生物可以利用来自电极的电子进行细胞内的分解反应和固碳作用[72],一些基于宏转录组的研究可以加深对这些过程中所涉及微生物的理解[73].一种未培养的可能的电自养菌Ca. Tenderia electrophaga在不同阴极电位下转录活性发生变化,相比其他物种,其在两种电位下具有更丰富的转录本,而相对活性与电流呈正相关.包括编码已知参与铁氧化的蛋白同源物的cyc2 基因在内的许多EET(extracellular electron transfer)关键组分的功能基因在470 mV下活跃表达,而在310 mV下二氧化碳固定相关基因的平均表达水平较高[73].而Marshall等[66]重建了阴极优势菌Acetobacterium、Sulfurospirillum和Desulfovibrio的代谢模型,发现Acetobacterium通过Wood-Ljungdahl途径将二氧化碳转化为乙酸,其在电极表面的水溶性氢化酶和铁氧还蛋白的转录本丰度高.然而在完全厌氧条件下,溶液中Sulfurospirillum的细胞色素C氧化酶却高度表达,需要后续研究来确定这些基因在电化学合成中的作用[66].
以基因组为中心的分析方法可以用于鉴定MES微生物群落中主要的EAB,并为EAB的生态生理学及其代谢作用提供了宝贵的信息.可以预见的是,今后更多的研究将会应用多组学的方法来深入理解MES功能微生物的代谢调控及种群互作机制.
3 总结与展望高通量核酸测序技术为研究自然和人工环境中未培养微生物的生理、代谢和进化等提供了工具,为揭示“微生物暗物质”的研究提供了方法和思路.微生物与微生物、微生物与生态环境的相互作用过程与机理仍然是微生物生态领域需要解决的重要问题.通过大数据的获取和生物信息学分析进行代谢重构与模拟,基于高通量核酸测序技术的宏基因组学、宏转录组学、基因蛋白质组学和单细胞基因组学将会被广泛应用于污水生物处理领域的研究.核酸提取作为宏组学研究的第一步,其提取效果对后续的测序会产生重要影响[74].环境样品的复杂性对DNA和RNA的提取带来了一定挑战,因此,选择合适的提取策略成为组学研究中的一大关键.随着单分子测序平台通量和准确性的提高,三代测序的组学研究将会得到广泛应用.便携式实时监测微生物群落的测序技术和单细胞捕获与单细胞基因组测序技术,将成为污水生物处理微生物种群研究的新领域.利用多组学和宏组学技术分析获取的种群互作和代谢调控机制,可以进一步指导反应器的设计、工程菌株开发、系统定向调控等,推动污水和废水处理技术的变革与发展.
| [1] |
FAUST K, RAES J. Microbial interactions: From networks to models[J]. Nature Reviews Microbiology, 2012, 10(8): 538. DOI:10.1038/nrmicro2832 |
| [2] |
WANG Z, ZHANG X, LU X, et al. Abundance and diversity of bacterial nitrifiers and denitrifiers and their functional genes in tannery wastewater treatment plants revealed by high-throughput sequencing[J]. PLoS One, 2014, 9(11): e113603. DOI:10.1371/journal.pone.0113603 |
| [3] |
STROUS M, PELLETIER E, MANGENOT S, et al. Deciphering the evolution and metabolism of an anammox bacterium from a community genome[J]. Nature, 2006, 440(7085): 790. DOI:10.1038/nature04647 |
| [4] |
SKENNERTON C T, BARR J J, SLATER F R, et al. Expanding our view of genomic diversity in Candidatus Accumulibacter clades[J]. Environmental Microbiology, 2015, 17(5): 1574. DOI:10.1111/1462-2920.12582 |
| [5] |
LIU Qian, REN Z J, HUANG Cong, et al. Multiple syntrophic interactions drive biohythane production from waste sludge in microbial electrolysis cells[J]. Biotechnology for Biofuels, 2016, 9: 162. DOI:10.1186/s13068-016-0579-x |
| [6] |
MEI Xiaoxue, XING Defeng, YANG Yang, et al. Adaptation of microbial community of the anode biofilm in microbial fuel cells to temperature[J]. Bioelectrochemistry, 2017, 117: 29. DOI:10.1016/j.bioelechem.2017.04.005 |
| [7] |
LIU Qian, LIU Bingfeng, LI Wei, et al. Impact of ferrous iron on microbial community of the biofilm in microbial fuel cells[J]. Frontiers in Microbiology, 2017, 8: 920. DOI:10.3389/fmicb.2017.00920 |
| [8] |
PARK H, ROSENTHAL A, JEZEK R, et al. Impact of inocula and growth mode on the molecular microbial ecology of anaerobic ammonia oxidation (anammox) bioreactor communities[J]. Water Research, 2010, 44(17): 5005. DOI:10.1016/j.watres.2010.07.022 |
| [9] |
PETERSON S B, WARNECKE F, MADEJSKA J, et al. Environmental distribution and population biology of Candidatus Accumulibacter, a primary agent of biological phosphorus removal[J]. Environmental Microbiology, 2008, 10(10): 2692. DOI:10.1111/j.1462-2920.2008.01690.x |
| [10] |
AMANN R I, LUDWIG W, SCHLEIFER K H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J]. Microbiological Reviews, 1995, 59(1): 143. |
| [11] |
RINKE C, SCHWIENTEK P, SCZYRBA A, et al. Insights into the phylogeny and coding potential of microbial dark matter[J]. Nature, 2013, 499(7459): 431. DOI:10.1038/nature12352 |
| [12] |
STEWART E J. Growing unculturable bacteria[J]. Journal of Bacteriology, 2012, 194(16): 4151. DOI:10.1128/JB.00345-12 |
| [13] |
孙欣, 高莹, 杨云锋. 环境微生物的宏基因组学研究新进展[J]. 生物多样性, 2013, 21(4): 393. SUN Xin, GAO Ying, YANG Yunfeng. Recent advancement in microbial environmental research using metagenomics tools[J]. Biodiversity Science, 2013, 21(4): 393. DOI:10.3724/SP.J.1003.2013.08050 |
| [14] |
韩丽丽, 吴娟, 马燕天, 等. 环境微生物转录组学研究进展[J]. 基因组学与应用生物学, 2017(12): 5210. HAN Lili, WU Juan, MA Yantian, et al. Advances in research on environmental microbial transcriptomics[J]. Genomics & Applied Biology, 2017(12): 5210. DOI:10.13417/j.gab.036.005210 |
| [15] |
HANDELSMAN J, RONDON M R, BRADY S F, et al. Molecular biological access to the chemistry of unknown soil microbes: A new frontier for natural products[J]. Chemistry & Biology, 1998, 5(10): R245. DOI:10.1016/S1074-5521(98)90108-9 |
| [16] |
ZHOU Jizhong, HE Zhili, YANG Yunfeng, et al. High-throughput metagenomic technologies for complex microbial community analysis: Open and closed formats[J]. MBio, 2015, 6(1): e2214. DOI:10.1128/mBio.02288-14 |
| [17] |
PORETSKY R S, BANO N, BUCHAN A, et al. Analysis of microbial gene transcripts in environmental samples[J]. Applied and Environmental Microbiology, 2005, 71(7): 4121. DOI:10.1128/AEM.71.7.4121-4126.2005 |
| [18] |
URICH T, LANZEN A, QI J, et al. Simultaneous assessment of soil microbial community structure and function through analysis of the meta-transcriptome[J]. PLoS One, 2008, 3(6): e2527. DOI:10.1371/journal.pone.0002527 |
| [19] |
RODRIGUEZ E, GARCIA-ENCINA P A, STAMS A J M, et al. Meta-omics approaches to understand and improve wastewater treatment systems[J]. Reviews in Environmental Science and Bio-Technology, 2015, 14(3): 385. DOI:10.1007/s11157-015-9370-x |
| [20] |
SANGER F, AIR G M, BARRELL B G, et al. Nucleotide sequence of bacteriophage φX174 DNA[J]. Nature, 1977, 265(5596): 687. DOI:10.1038/265687a0 |
| [21] |
FLEISCHMANN R D, ADAMS M D, WHITE O, et al. Whole-genome random sequencing and assembly of Haemophilus influenzae Rd[J]. Science, 1995, 269(5223): 496. DOI:10.1126/science.7542800 |
| [22] |
COLLINS F S, MORGAN M, PATRINOS A. The human genome project: Lessons from large-scale biology[J]. Science, 2003, 300(5617): 286. DOI:10.1126/science.1084564 |
| [23] |
MARGULIES M, EGHOLM M, ALTMAN W E, et al. Genome sequencing in microfabricated high-density picolitre reactors[J]. Nature, 2005, 437(7057): 376. DOI:10.1038/nature03959 |
| [24] |
SHENDURE J, PORRECA G J, REPPAS N B, et al. Accurate multiplex polony sequencing of an evolved bacterial genome[J]. Science, 2005, 309(5741): 1728. DOI:10.1126/science.1117389 |
| [25] |
BENTLEY D R, BALASUBRAMANIAN S, SWERDLOW H P, et al. Accurate whole human genome sequencing using reversible terminator chemistry[J]. Nature, 2008, 456(7218): 53. DOI:10.1038/nature07517 |
| [26] |
Di BELLA J M, BAO Y, GLOOR G B, et al. High throughput sequencing methods and analysis for microbiome research[J]. Journal of Microbiological Methods, 2013, 95(3): 401. DOI:10.1016/j.mimet.2013.08.011 |
| [27] |
EID J, FEHR A, GRAY J, et al. Real-time DNA sequencing from single polymerase molecules[J]. Science, 2009, 323(5910): 133. DOI:10.1126/science.1162986 |
| [28] |
BRANTON D, DEAMER D W, MARZIALI A, et al. The potential and challenges of nanopore sequencing[J]. Nature Biotechnology, 2008, 26(10): 1146. DOI:10.1038/nbt.1495 |
| [29] |
叶姜瑜, 罗固源, 吉芳英, 等. 污水生物处理功能微生物的多样性[J]. 重庆大学学报(自然科学版), 2005, 28(10): 119. YE Jiangyu, LUO Guyuan, JI Fangying, et al. Functional microbial diversities in wastewater biological treatment[J]. Journal of Chongqing University (Natural Science Edition), 2005, 28(10): 119. |
| [30] |
SANGWAN N, XIA F, GILBERT J A. Recovering complete and draft population genomes from metagenome datasets[J]. Microbiome, 2016, 4: 8. DOI:10.1186/s40168-016-0154-5 |
| [31] |
YU K, ZHANG T. Metagenomic and metatranscriptomic analysis of microbial community structure and gene expression of activated sludge[J]. PLoS One, 2012, 7(5): e38183. DOI:10.1371/journal.pone.0038183 |
| [32] |
ISHⅡ S, SUZUKI S, NORDEN-KRICHMAR T M, et al. A novel metatranscriptomic approach to identify gene expression dynamics during extracellular electron transfer[J]. Nature Communications, 2013, 4: 1601. DOI:10.1038/ncomms2615 |
| [33] |
HE S, KUNIN V, HAYNES M, et al. Metatranscriptomic array analysis of 'Candidatus Accumulibacter phosphatis'-enriched enhanced biological phosphorus removal sludge[J]. Environmental Microbiology, 2010, 12(5): 1205. DOI:10.1111/j.1462-2920.2010.02163.x |
| [34] |
KUYPERS M M M, MARCHANT H K, KARTAL B. The microbial nitrogen-cycling network[J]. Nature Reviews Microbiology, 2018, 16(5): 263. DOI:10.1038/nrmicro.2018.9 |
| [35] |
TIAN Mei, ZHAO Fangqing, SHEN Xin, et al. The first metagenome of activated sludge from full-scale anaerobic/anoxic/oxic (A2O) nitrogen and phosphorus removal reactor using Illumina sequencing[J]. Journal of Environmental Sciences, 2015, 35: 181. DOI:10.1016/j.jes.2014.12.027 |
| [36] |
YE Lin, ZHANG Tong, WANG Taitao, et al. Microbial structures, functions, and metabolic pathways in wastewater treatment bioreactors revealed using high-throughput sequencing[J]. Environmental Science & Technology, 2012, 46(24): 13244. DOI:10.1021/es303454k |
| [37] |
MIAO Yu, LIAO Runhua, ZHANG Xuxiang, et al. Metagenomic insights into Cr(VI) effect on microbial communities and functional genes of an expanded granular sludge bed reactor treating high-nitrate wastewater[J]. Water Research, 2015, 76: 43. DOI:10.1016/j.watres.2015.02.042 |
| [38] |
MIAO Yu, LIAO Runhua, ZHANG Xuxiang, et al. Metagenomic insights into salinity effect on diversity and abundance of denitrifying bacteria and genes in an expanded granular sludge bed reactor treating high-nitrate wastewater[J]. Chemical Engineering Journal, 2015, 277: 116. DOI:10.1016/j.cej.2015.04.125 |
| [39] |
HU Z, SPETH D R, FRANCOIJS K, et al. Metagenome analysis of a complex community reveals the metabolic blueprint of anannmox bacterium "Candidatus Jettenia asiatica"[J]. Frontiers in Microbiology, 2012, 3: 366. DOI:10.3389/fmicb.2012.00366 |
| [40] |
TANG Xi, GUO Yongzhao, JIANG Bo, et al. Metagenomic approaches to understanding bacterial communication during the anammox reactor start-up[J]. Water Research, 2018, 136: 95. DOI:10.1016/j.watres.2018.02.054 |
| [41] |
SPETH D R, INT ZANDT M H, GUERRERO-CRUZ S, et al. Genome-based microbial ecology of anammox granules in a full-scale wastewater treatment system[J]. Nature Communications, 2016, 7: 11172. DOI:10.1038/ncomms11172 |
| [42] |
GUO Jianhua, PENG Yongzhen, FAN Lu, et al. Metagenomic analysis of anammox communities in three different microbial aggregates[J]. Environmental Microbiology, 2016, 18(9): 2979. DOI:10.1111/1462-2920.13132 |
| [43] |
BHATTACHARJEE A S, WU S, LAWSON C E, et al. Whole-community metagenomics in two different anammox configurations: Process performance and community structure[J]. Environmental Science & Technology, 2017, 51(8): 4317. DOI:10.1021/acs.est.6b05855 |
| [44] |
LAWSON C E, WU S, BHATTACHARJEE A S, et al. Metabolic network analysis reveals microbial community interactions in anammox granules[J]. Nature Communications, 2017, 8: 15416. DOI:10.1038/ncomms15416 |
| [45] |
GONZALEZ-MARTINEZ A, MARGARETO A, RODRIGUEZ-SANCHEZ A, et al. Linking the effect of antibiotics on partial-nitritation biofilters: Performance, microbial communities and microbial activities[J]. Frontiers in Microbiology, 2018, 9: 354. DOI:10.3389/fmicb.2018.00354 |
| [46] |
SALES C M, LEE P K H. Resource recovery from wastewater: Application of meta-omics to phosphorus and carbon management[J]. Current Opinion in Biotechnology, 2015, 33: 260. DOI:10.1016/j.copbio.2015.03.003 |
| [47] |
MARTIN H G, IVANOVA N, KUNIN V, et al. Metagenomic analysis of two enhanced biological phosphorus removal (EBPR) sludge communities[J]. Nature Biotechnology, 2006, 24(10): 1263. DOI:10.1038/nbt1247 |
| [48] |
HE S, GALL D L, MCMAHON K D. "Candidatus accumulibacter" population structure in enhanced biological phosphorus removal sludges as revealed by polyphosphate kinase genes[J]. Applied and Environmental Microbiology, 2007, 73(18): 5865. DOI:10.1128/AEM.01207-07 |
| [49] |
HESSELMANN R, WERLEN C, HAHN D, et al. Enrichment, phylogenetic analysis and detection of a bacterium that performs enhanced biological phosphate removal in activated sludge[J]. Systematic and Applied Microbiology, 1999, 22(3): 454. DOI:10.1016/S0723-2020(99)80055-1 |
| [50] |
FLOWERS J J, HE S, MALFATTI S, et al. Comparative genomics of two 'Candidatus Accumulibacter' clades performing biological phosphorus removal[J]. ISME Journal, 2013, 7(12): 2301. DOI:10.1038/ismej.2013.117 |
| [51] |
OEHMEN A, LEMOS P C, CARVALHO G, et al. Advances in enhanced biological phosphorus removal: From micro to macro scale[J]. Water Research, 2007, 41(11): 2271. DOI:10.1016/j.watres.2007.02.030 |
| [52] |
MCILROY S J, ALBERTSEN M, ANDRESEN E K, et al. 'Candidatus Competibacter'-lineage genomes retrieved from metagenomes reveal functional metabolic diversity[J]. ISME Journal, 2014, 8(3): 613. DOI:10.1038/ismej.2013.162 |
| [53] |
ALBERTSEN M, MCILROY S J, STOKHOLM-BJERREGAARD M, et al. "Candidatus Propionivibrio aalborgensis": A novel glycogen accumulating organism abundant in full-scale enhanced biological phosphorus removal plants[J]. Frontiers in Microbiology, 2016, 7: 1033. DOI:10.3389/fmicb.2016.01033 |
| [54] |
ALBERTSEN M, HANSEN L B S, SAUNDERS A M, et al. A metagenome of a full-scale microbial community carrying out enhanced biological phosphorus removal[J]. ISME Journal, 2012, 6(6): 1094. DOI:10.1038/ismej.2011.176 |
| [55] |
COKRO A A, LAW Y, WILLIAMS R B H, et al. Non-denitrifying polyphosphate accumulating organisms obviate requirement for anaerobic condition[J]. Water Research, 2017, 111: 393. DOI:10.1016/j.watres.2017.01.006 |
| [56] |
MAO Y, YU K, XIA Y, et al. Genome reconstruction and gene expression of "Candidatus Accumulibacter phosphatis" Clade IB performing biological phosphorus removal[J]. Environmental Science & Technology, 2014, 48(17): 10363. DOI:10.1021/es502642b |
| [57] |
OYSERMAN B O, NOGUERA D R, DEL RIO T G, et al. Metatranscriptomic insights on gene expression and regulatory controls in Candidatus Accumulibacter phosphatis[J]. ISME Journal, 2016, 10(4): 810. DOI:10.1038/ismej.2015.155 |
| [58] |
LOPEZ-VAZQUEZ C M, OEHMEN A, HOOIJMANS C M, et al. Modeling the PAO-GAO competition: Effects of carbon source, pH and temperature[J]. Water Research, 2009, 43(2): 450. DOI:10.1016/j.watres.2008.10.032 |
| [59] |
LAW Y, KIRKEGAARD R H, COKRO A A, et al. Integrative microbial community analysis reveals full-scale enhanced biological phosphorus removal under tropical conditions[J]. Scientific Reports, 2016, 6: 25719. DOI:10.1038/srep25719 |
| [60] |
MEI Xiaoxue, GUO Changhong, LIU Bingfeng, et al. Shaping of bacterial community structure in microbial fuel cells by different inocula[J]. RSC Advances, 2015, 5(95): 78136. DOI:10.1039/c5ra16382j |
| [61] |
ISHⅡ S, SUZUKI S, NORDEN-KRICHMAR T M, et al. Microbial population and functional dynamics associated with surface potential and carbon metabolism[J]. ISME Journal, 2014, 8(5): 963. DOI:10.1038/ismej.2013.217 |
| [62] |
KISELEVA L, GARUSHYANTS S K, MA H, et al. Taxonomic and functional metagenomic analysis of anodic communities in two pilot-scale microbial fuel cells treating different industrial wastewaters[J]. Journal of Integrative Bioinformatics, 2015, 12(3): 273. DOI:10.2390/biecoll-jib-2015-273 |
| [63] |
ZHANG Husen, CHEN Xi, BRAITHWAITE D, et al. Phylogenetic and metagenomic analyses of substrate-dependent bacterial temporal dynamics in microbial fuel cells[J]. PLoS One, 2014, 9(9): e107460. DOI:10.1371/journal.pone.0107460 |
| [64] |
KOUZUMA A, KASAI T, NAKAGAWA G, et al. Comparative metagenomics of anode-associated microbiomes developed in rice paddy-field microbial fuel cells[J]. PLoS One, 2013, 8(11): e77443. DOI:10.1371/journal.pone.0077443 |
| [65] |
YAMAMURO A, KOUZUMA A, ABE T, et al. Metagenomic analyses reveal the involvement of syntrophic consortia in methanol/electricity conversion in microbial fuel cells[J]. PLoS One, 2014, 9(5): e98425. DOI:10.1371/journal.pone.0098425 |
| [66] |
MARSHALL C W, ROSS D E, HANDLEY K M, et al. Metabolic reconstruction and modeling microbial electrosynthesis[J]. Scientific Reports, 2017, 7: 8391. DOI:10.1038/s41598-017-08877-z |
| [67] |
ROSS D E, MARSHALL C W, MAY H D, et al. Draft genome sequence of Sulfurospirillum sp. strain MES, reconstructed from the metagenome of a microbial electrosynthesis system[J]. Genome Announcements, 2015, 3(1). DOI:10.1128/genomeA.01336-14 |
| [68] |
WANG Z, LEARY D H, MALANOSKI A P, et al. A previously uncharacterized, nonphotosynthetic member of the Chromatiaceae is the primary CO2-fixing constituent in a self-regenerating biocathode[J]. Applied and Environmental Microbiology, 2015, 81(2): 699. DOI:10.1128/AEM.02947-14 |
| [69] |
EDDIE B J, WANG Z, MALANOSKI A P, et al. 'Candidatus Tenderia electrophaga', an uncultivated electroautotroph from a biocathode enrichment[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(6): 2178. DOI:10.1099/ijsem.0.001006 |
| [70] |
MALANOSKI A P, LIN B, EDDIE B J, et al. Relative abundance of "Candidatus Tenderia electrophaga" is linked to cathodic current in an aerobic biocathode community[J]. Microbial Biotechnology, 2018, 11(1): 98. DOI:10.1111/1751-7915.12757 |
| [71] |
ISHⅡ S, SUZUKI S, TENNEY A, et al. Microbial metabolic networks in a complex electrogenic biofilm recovered from a stimulus-induced metatranscriptomics approach[J]. Scientific Reports, 2015, 5: 14840. DOI:10.1038/srep14840 |
| [72] |
TREMBLAY P, ZHANG T. Electrifying microbes for the production of chemicals[J]. Frontiers in Microbiology, 2015, 6: 201. DOI:10.3389/fmicb.2015.00201 |
| [73] |
EDDIE B J, WANG Z, HERVEY W J, et al. Metatranscriptomics supports the mechanism for biocathode electroautotrophy by "Candidatus Tenderia electrophaga"[J]. MSystems, 2017, 2(2): e2. DOI:10.1128/mSystems.00002-17 |
| [74] |
COSTEA P I, ZELLER G, SUNAGAWA S, et al. Towards standards for human fecal sample processing in metagenomic studies[J]. Nature Biotechnology, 2017, 35(11): 1069. DOI:10.1038/nbt.3960 |
 2019, Vol. 51
2019, Vol. 51


