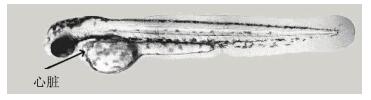
斑马鱼是一种基因序列与人类具有较高的相似性的有脊椎模式动物[1],其幼鱼具有透明表皮,能够直接观察内脏器官的发育以及血液循环系统的运行情况(如图 1所示[2]),是生物医学领域的重要实验对象[3].2019年,伯尔尼大学揭示了斑马鱼幼鱼心脏组织自我再生能力的机理,引发了国际上对斑马鱼幼鱼心血管系统的广泛研究[4].对幼鱼心脏的识别与监测对于理解其发育机理[5]、药物影响[6]、组织再生[7]等过程至关重要.但是,由于斑马鱼幼鱼体长仅有3 mm左右,其内部器官细小、各异性、色素血管等的干扰,以往的人工观察手段在准确性和效率上都有很大不足.如对于幼鱼心脏观测来说,幼鱼需要处于特定姿态(如侧躺)才能够使心脏暴露在显微镜视野下方,其心脏直径约为100 μm,对于人工和常规的显微操作系统来说存在较大困难.
|
图 1 48~72 hpf斑马鱼幼鱼形态示例 Fig. 1 Example of zebrafish larva at 48~72 hpf |
针对上述问题,本文基于已搭建的斑马鱼幼鱼显微注射平台[8]开展斑马鱼幼鱼心脏识别和监测方法的研究.该方法在多条幼鱼放入平台托盘后,利用显微镜获取幼鱼在托盘中的分布图像,并基于高斯混合模型对幼鱼进行分割、定位;利用卷积神经网络对幼鱼的姿态进行识别,并通过带有吸持针的滚转机械臂将幼鱼滚转角调整至统一姿态;逐一对幼鱼进行高倍率显微观察,得到幼鱼心脏图像,并通过像素强度跟踪算法,获取并监测幼鱼心率.该方法能实现高通量的斑马鱼幼鱼心脏进行准确识别和在线检测,能够帮助生物实验人员快速获得实验数据,提高研究效率和水平.
1 系统设计本文基于图 2展示的斑马鱼幼鱼显微操作系统[9].该系统以电动体式显微镜(Nikon-SMZ25)为基础,顶部CMOS相机最大帧率为60帧/s.底座改装为带有旋转托盘和三自由度机械臂的机械平台,机械臂末端连接带有旋转功能的玻璃吸持针用于幼鱼姿态控制.该图像处理及控制软件在VS2010上采用MFC编写.

|
图 2 斑马鱼显微操作系统 Fig. 2 Micromanipulation system for zebrafish larvae |
在观察实验开始,多条经MS-222(质量分数为0.003%)麻醉后的年龄范围为48~120 hpf的幼鱼被一次性放入托盘中,并使用低倍放大倍率进行图像采集.为利用高倍率物镜对每只幼鱼进行心脏监测,需要对图像中的幼鱼进行分割定位,并进行姿态调整到易于观测的位置.本文基于高斯混合模型[10],实现对多条幼鱼进行分割定位.幼鱼本身具有较为明显的身体特征,例如眼睛和鱼鳔等具有较深的颜色信息,因此利用二值化方法,可以将其从背景中分割出来,如图 3(b)所示.

|
图 3 斑马鱼幼鱼分割定位 Fig. 3 Segmentation and location of zebrafish larvae |
为了能确定每个像素点所属的斑马鱼幼鱼以及获得斑马鱼幼鱼的位置,将经二值化后的像素点看作X∈Nn×2的数据集,使用高斯混合模型分类.对于含有K类的高斯混合模型,对每一类的参数采用EM算法进行迭代计算:
| $ \left\{ \begin{align} & {{\omega }_{j}}=\frac{1}{n}\sum\limits_{i=1}^{n}{{{\gamma }_{j}}({{x}_{i}}), } \\ & {{\mu }_{j}}=\frac{\sum\limits_{i=1}^{n}{{{\gamma }_{j}}({{x}_{i}}){{x}_{i}}}}{\sum\limits_{i=1}^{n}{{{\gamma }_{j}}({{x}_{i}})}}, \\ & {{\sum }_{j}}=\frac{\sum\limits_{i=1}^{n}{{{\gamma }_{j}}({{x}_{i}})({{x}_{i}}-{{\mu }_{j}}){{({{x}_{i}}-{{\mu }_{j}})}^{\text{T}}}}}{\sum\limits_{i=1}^{n}{{{\gamma }_{j}}({{x}_{i}})}}. \\ \end{align} \right. $ |
式中:
| $ {{J}_{l+1}}-{{J}_{l}}>-\bar{J}, l\in {{N}_{+}} $ |
式中,J为由经验求出的算法终止迭代阈值.在使用该方法的时会出现将一条幼鱼的像素分为两类,以及多条幼鱼分为一类的情况,这是由于模型初始值所确定的均值μk相近,和幼鱼重叠造成的.为避免该问题,每次迭代开始前应将初值位置尽量分隔开,以及均匀摆放幼鱼使每条幼鱼相互间位置尽可能大.对于如图 3(a)所示的幼鱼,其经高斯混合模型后所得参数所绘制的曲面图如图 3(c)所示.图 3(d)显示了根据获得的均值μk确定的每幼鱼的位置.
由于幼鱼双眼和鱼鳔处颜色较深,因此能定位到该位置即能成功找到幼鱼.在稳定的光线条件下,双眼和鱼鳔处与背景对比度高,因此该方法能够准确实现幼鱼的分割及定位.
2.2 幼鱼二维姿态检测与控制对于麻醉后的斑马鱼幼鱼,其在水中的自然姿态大部分为侧躺姿态,因此心脏能够进入显微镜视野,但是也有部分幼鱼姿态会出现鱼肚朝下或朝上的异常姿态(如图 3(a)中箭头标识).为便于后续对心脏的观测,需要调整斑马鱼姿态使心脏成像清晰完整.本系统利用旋转托盘和吸持机械臂实现幼鱼姿态调整.在这一过程中,首先根据上一步确定的幼鱼位置时获得的高斯模型参数,对待观测的幼鱼,根据其高斯模型的协方差估计其当前二维姿态.
对于二维姿态的确定,由图 3(a)所知,斑马鱼身体细长,因此对于一条鱼的所有像素点组成的二维数据集D∈Nn×2,其方差最大的方向v可定义为该幼鱼的当前姿态.类似于主成分分析法,由数据的协方差矩阵
| $ \begin{align} & {{\sum }_{\text{pro}}}=\frac{1}{N}{{\sum\limits_{i=1}^{n}{\left( {{\left( {{x}_{i}}-{{m}_{i}} \right)}^{\text{T}}}v \right)}}^{2}}= \\ & \frac{1}{N}\sum\limits_{i=1}^{n}{{{v}^{T}}\left( {{x}_{i}}-{{m}_{i}} \right)}{{({{x}_{i}}-{{m}_{i}})}^{\text{T}}}v={{v}^{\text{T}}}\sum v \\ \end{align} $ |
则目标为所求方向向量v使Σpro最大.由于Σ为Hermitian矩阵,且vTv=1,则vTΣv为瑞利熵.由其性质可知,当瑞利熵取最大值时,v为Σ最大特征值对应的特征向量.因此根据每个样本协方差矩阵Σk的特征向量能够获得每条斑马鱼幼鱼二维姿态vk.为了使吸持机械臂能够捕捉到幼鱼尾部,因此将所求高斯模型的协方差求取其最大特征值所对应的特征向量作为幼鱼方向,例如图 3(a)中,所得各个幼鱼的方向如图 4(d)所示.定义所得到的高斯模型的均值点与尾部端点的连线代表尾部的朝向,其与协方差求得的方向之差代表旋转的误差.经实验测试,该方法所得误差分布在±10°之间,不影响对斑马鱼尾部的吸持.
获取二维姿态后由上位机旋转托盘,转到尾部朝向旋转机械臂的位置,定位针尖位置并移动旋转机械臂,吸住斑马鱼幼鱼尾部,将幼鱼调整二维位置至视野中心,进行滚转角调整[11].
2.3 幼鱼滚转角的检测控制在吸住尾部后,需要对幼鱼的滚转角进行调整,以使心脏处于最佳观测位置.本文搭建了卷积神经网络对幼鱼当前角度进行估计,并控制滚转轴对幼鱼进行调整.
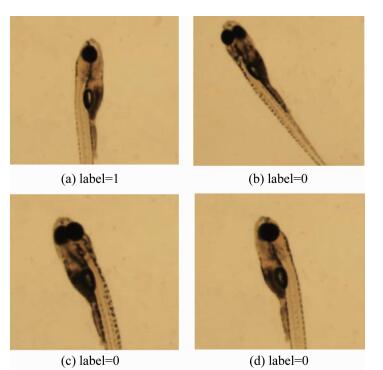
在卷积神经网络的搭建过程中,最重要的一步是样本集的设计与建立.结合系统中对滚转角判断的需要,存在两种可用的样本集标注方式,分别为直接角度标注和区域分类标注.直接角度标注是指对应每一幅样本图片,其对应的标签为视野中的幼鱼相对于某一基准的偏离角度;而区域分类标注则是指当幼鱼的滚转角处于某一有利于心脏观测的姿态时(如幼鱼侧放且心脏朝向水平向右),标记为合格样本,其余姿态为不合格样本.由于在单目视觉条件下,难以对精确角度进行测量,因此选用第2种标注方法.相比之下这种方法具有以下优点:1)由于在标注过程中姿态判断是基于人工主观标记,因此标签集中对于角度限制较为宽松,对于姿态调整降低了要求,同时简化了标签集的制作工作. 2)卷积神经网络在解决分类问题时能够对人工判断经验进行学习,使分类结果得更为稳定,因此使用判断角度范围会增加系统的鲁棒性能.使用该方法的不同姿态所对应的标签如图 4所示.
|
图 4 不同斑马鱼幼鱼的姿态及对应标签 Fig. 4 Postures of zebrafish larvae and corresponding labels |
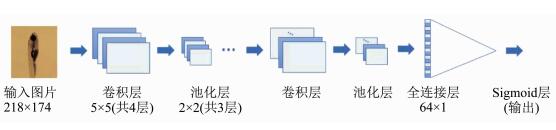
网络搭建从浅层卷积神经网络开始,逐渐增加网络层数,通过实验结果来确定最佳的网络结构.经过测试,网络中采用3×3的卷积核时能够取得比较理想的训练效果.为了尽可能降低运算复杂程度,同时避免优化过程中的梯度爆炸与梯度消失问题,激活函数选取ReLU函数[12].在优化方法的选择上,Adam方法收敛速度相对较快,能够缩短训练时间[13].由于所选取的网络层数较深,为降低过拟合风险,网络内部加入了dropout和池化层[14].最终的预测数据通过全连接层输出,得到的结果即为幼鱼正放的概率.首先需将输入图片尺寸压缩为218×174,整个网络包含输入层、4层卷积层、3层池化层和1层全连接层,结构如图 5所示.训练集由50条处于1~5 dpf内不同年龄段的幼鱼组成.每条幼鱼以1°为步长旋转360°,当幼鱼处于人工判别为可观测心脏的角度范围内时,对应标签记为1,其余状态记为0.在训练过程中,当精度达到99%时认为算法收敛,训练停止.按照相同的方法,将另外10条幼鱼的数据作为测试集输入网络.训练过程在迭代15次后结束,测试结果准确度约为99.4%.
|
图 5 滚转角检测的卷积神经网络结构 Fig. 5 Convolutional neural network structure of roll angle detection |
在对目标幼鱼进行滚转调整时,首先使用旋转机械臂吸住幼鱼尾部旋转1周,并以1°为步长,采集360张样本图,将这些图放入训练好的模型进行样本图片的预测,记录其最佳角度的概率值,做为该张图片处在最适合注射的空间位置,控制机械臂反向旋转,使幼鱼旋转到上述所对应的空间角度,即为目标滚转角.
本文采用峰值旁瓣比(PSR)指标[15]对预测斑马鱼最佳观测姿态的卷积神经网络进行评估.对于预测斑马鱼最佳姿态,输出值的波峰出现在幼鱼心脏处于右侧时.随着幼鱼滚转,输出值逐渐减小.假设选择波峰窗口为30°,则PSR的计算方法为:幼鱼滚转1周的波峰/除去幼鱼心脏恰好处于右侧±15°后的波峰.当所得PSR越大时,代表算法越稳定.本文对多条幼鱼翻滚滚转1周进行试验,并由此计算PSR,其大小为70%,因此该算法稳定.
3 斑马鱼心率监测 3.1 幼鱼心脏定位在对幼鱼姿态进行调整后,切换显微镜放大倍率,将相机的图像采样频率设定为50帧/s,对单个幼鱼心脏进行观察.由于存在环境噪声以及幼鱼新陈代谢活动对心脏监测算法的干扰和影响,需要首先对幼鱼的心脏进行识别和定位.斑马鱼幼鱼血细胞会集中在心脏位置且在显微镜下呈红色,并且在相同成像条件下相同品系和年龄的幼鱼成像效果是近似的,因此可以将包含幼鱼的RGB图像转化到HSV空间,再提取图像中明度较高的红色区域,根据实验测试,本文取-30° < H < 30°且V>50%作为阈值.由于斑马鱼的血细胞几乎分布在身体的各个区域,因此需要对高明度红色区域进行开运算,取其中面积最大区域作为心脏位置,并建立矩形心脏观测区域(HR)作为下一步的心跳监测区域,建立过程如图 6所示.

|
图 6 心脏区域的提取 Fig. 6 Extraction of heart region |
由于心脏定位的算法根据心脏颜色特征进行识别定位,因此该方法较容易受身体部位色素覆盖了心脏的颜色特征的影响,使得所定位面积比心脏真实区域小.对此本文是根据所求得的心脏区域的重心作为一个矩形中心,在矩形中进一步进行心率识别.虽然该方法会引入部分噪声,但对后续影响较小.
3.2 斑马鱼幼鱼心率监测在定位到心脏区域后,通过如下的像素强度跟踪算法来实现心率的检测.其以图像差分为基本方法.但是由于水流等因素的影响,幼鱼整体可能存在缓慢的漂移,这会导致在差分图像中始终存在噪声,为接下来的心跳检测带来了不确定性.为了抑制噪声,首先对原始图像中的心脏区域进行高斯滤波.但是常规的相邻帧差分方法以及相对固定帧差分均无法较好地反映检测心脏的运动变化,因此本文将上一步中得到的HR区域灰度化并取每个像素在超过一个心跳周期的时间中取到的最大值,以此作为差分法的参考图像.得到的心脏区域内一个心跳周期内的强度变化如图 7所示.

|
图 7 一个心跳周期内心脏区域像素强度变化 Fig. 7 Changes of pixel intensity of heart region in a heartbeat cycle |
从图 7中可以观察到,心脏区域内强度变化的重心位置随着血液在心室心房中的流动而变化,因此可通过跟踪像素强度变化的重心位置实现对心跳的监测,其计算方式如下:
| $ G=\left( {{x}^{\text{C}}}, {{y}^{\text{C}}} \right)=\left( \frac{{{M}_{10}}}{{{M}_{00}}}, \frac{{{M}_{01}}}{{{M}_{00}}} \right). $ |
式中:G为重心位置; (xC, yC)为重心坐标;
为了将重心在二维图像上的变化转化为单一指标来描述心跳,将重心位置坐标(x, y)向其方差最大的方向(a, b)投影,即
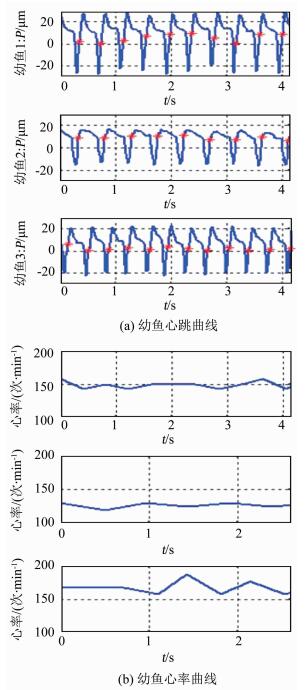
图 8(a)为采用上述像素强度跟踪方法对3条不同幼鱼进行的心跳监测结果.图中曲线由心脏的重心随时间而发生的位移构成.通过人工逐帧对幼鱼心脏血液变化进行判断,并与算法结果进行比较,两者差距一般在20 μm内,能够反映心脏的跳动规律,其波动曲线能够帮助对幼鱼生理状态进行研究.图中红色星号为心率周期标记,可以得到3条幼鱼的心率曲线,如图 8(b)所示,可以看出每条幼鱼自身的心率变化都围绕着150次/min变化,与文献[16]中的研究结果相吻合.
|
图 8 不同幼鱼在一段时间内的心脏监测 Fig. 8 Heart rate detection of zebrafish larvae in a period of time |
1) 本文基于高斯混合模型确定多条斑马鱼幼鱼的二维姿态和在视野中的位置,基于卷积神经网络确定幼鱼翻滚角,以此实现了幼鱼的位姿检测和调整控制.
2) 根据斑马鱼幼鱼心脏的特点,提出了一种基于像素强度跟踪的周期性运动目标识别方法,能够定位心脏区域,并实现心跳曲线的高效监测.
3) 本文基于显微视觉实现了高通量的斑马鱼幼鱼心脏识别和监测方法,该方法无需植入电极或注射荧光物质,能够保证幼鱼在最小损伤及改造的条件下监测幼鱼状态.为有脊椎动物的遗传、发育、组织重生的研究提供了一种新颖的高通量手段,同时也为提高心脏类药物的研发和测试效率提供参考.
| [1] |
HOWE K, CLARK M D, TORROJA C F, et al. The zebrafish reference genome sequence and its relationship to the human genome[J]. Nature, 2013, 496(7446): 498. DOI:10.1038/nature12111 |
| [2] |
GRAHAM K. Biology development[EB/OL]. [2020-03-01].https://msu.edu/~bentle48/Katie%20Graham.html.
|
| [3] |
COSENTINO C C, ROMAN B L, DRUMMOND I A, et al. Intravenous microinjections of zebrafish larvae to study acute kidney injury[J]. Journal of Visualized Experiments, 2010, 12(42): e2079. DOI:10.3791/2079(2010) |
| [4] |
SANDE M, MARCOS M, INES G, et al. Adult sox10+ cardiomyocytes contribute to myocardial regeneration in the zebrafish[J]. Cell Reports, 2019, 29(4): 1041. DOI:10.1016/j.celrep.2019.09.041 |
| [5] |
KAWAHARA A, NISHI T, HISANO Y, et al. The sphingolipid transporter Spns2 functions in migration of zebrafish myocardial precursors[J]. Science, 2009, 323(5913): 524. DOI:10.1126/science.1167449 |
| [6] |
ZHU Xiaoyu, LIU Hongcui, GUO Shengya, et al. A zebrafish thrombosis model for assessing antithrombotic drugs[J]. Zebrafish, 2016, 13(4): 335. DOI:10.1089/zeb.2016.1263 |
| [7] |
D'AMICO L, SCOTT I C, JUNGBLUT B, et al. A mutation in zebrafish hmgcr1b reveals a role for isoprenoids in vertebrate heart-tube formation[J]. Current Biology, 2007, 17(3): 252. DOI:10.1016/j.cub.2006.12.023 |
| [8] |
庄松霖.面向斑马鱼幼鱼的显微操作系统关键技术研究[D].哈尔滨: 哈尔滨工业大学, 2019 ZHUANG Songlin. Research on key technologies of zebrafish larva micromanipulation system[D]. Harbin: Harbin Institute of Technology, 2019 |
| [9] |
ZHUANG Songlin, LIN Weiyang, ZHONG Jie, et al. Visual servoed three-dimensional rotation control in zebrafish larva heart microinjection system[J]. IEEE Transactions on Biomedical Engineering, 2018, 65(1): 64. DOI:10.1109/TBME.2017.2688375 |
| [10] |
周志华. 机器学习[M]. 北京: 清华大学出版社, 2016: 162. ZHOU Zhihua. Machine learning[M]. Beijing: Tsinghua University Press, 2016: 162. |
| [11] |
ZHUANG Songlin, LIN Weiyang, GAO Huijun, et al. Visual servoed zebrafish larva heart microinjection system[J]. IEEE Transactions on Industrial Electronics, 2017, 64(5): 3727. DOI:10.1109/TIE.2017.2652380 |
| [12] |
HAHNLOSER R H R, SARPESHKAR R, MAHOWALD M A, et al. Digital selection and analogue amplification coexist in a cortex-inspired silicon circuit[J]. Nature, 2000, 405(6789): 947. DOI:10.1038/35016072 |
| [13] |
KHAN S H, RAHMANI H, SHAH S A A, et al. A guide to convolutional neural networks for computer vision[J]. Synthesis Lectures on Computer Vision, 2018, 8(1): 1. DOI:10.2200/S00822ED1V01Y201712COV015 |
| [14] |
LECUN Y, BENGIO Y, HINTON G. Deep learning[J]. Nature, 2015, 521(7553): 436. DOI:10.1038/nature14539 |
| [15] |
JUNG C H, KWAG Y K. PSLR estimation considering clutter background from SAR image data[C]//Proceedings of the IEEE International Geoscience & Remote Sensing Symposium. Piscataway, NJ: IEEE Press, 2009: 627. DOI: 10.1109/IGARSS.2009.5417447
|
| [16] |
KRISHNA S, CHATTI K, GALIGEKERE R R. Automatic and robust estimation of heart rate in zebrafish larvae[J]. IEEE Transactions on Automation Science & Engineering, 2018, 15(3): 1041. DOI:10.1109/TASE.2017.2705240 |
 2020, Vol. 52
2020, Vol. 52


