2. 水质科学与水环境恢复工程北京市重点实验室(北京工业大学), 100124 北京;
3. 中国建筑科学研究院建筑设计院, 100013 北京;
4. 城市水资源与水环境国家重点实验室(哈尔滨工业大学), 150090 哈尔滨
2. Key Laboratory of Beijing for Water Quality Science and Water Environment Recovery Engineering(Beijing University of Technology), 100124 Beijing, China;
3. Architectural Design Institute, China Academy of Building Research, 100013 Beijing, China;
4. State Key Laboratory of Urban Water Resource and Environment(Harbin Institute of Technology), 150090 Harbin, China
传统生物脱氮工艺经过硝化和反硝化作用两个阶段完成脱氮过程,硝化反应包括氨氧化与亚硝酸盐氧化两个过程,分别由氨氧化菌(AOB)和亚硝酸盐氧化菌(NOB)负责完成.亚硝化过程就是使硝化反应停留在氨氧化阶段,避免亚硝酸盐被氧化成硝酸盐,从而形成亚硝酸盐积累.若亚硝化过程与后续厌氧氨氧化工艺耦合(PN-ANAMMOX),则只需要将57%的氨氧化成亚硝酸盐,剩余的氨氮与亚硝酸盐按如下反应式完成脱氮过程[1],即
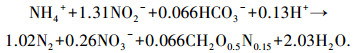
|
与传统硝化/反硝化脱氮作用相比,PN-ANAMMOX理论上可节省62.5%的曝气能耗,无需有机碳源,且污泥产量低,受到广泛关注[2].若要维持常温低基质生活污水PN-ANAMMOX工艺稳定运行,关键在于实现稳定的亚硝化过程,即PN反应器出水NO2-与NH4+比值稳定,实质就是控制条件促进AOB生长繁殖而淘汰NOB[3].本研究以经过厌氧-好氧(A/O)除磷工艺去除磷及大部分有机物的生活污水为对象,完成亚硝化(PN)反应器的启动.待PN反应器稳定运行后,通过扫描电镜(SEM)、荧光原位杂交(FISH)、变性梯度凝胶电泳(DGGE)等技术,分析污泥微观结构、解析功能菌群特性,探索功能菌群与反应器亚硝化效果之间的关系,为促进AOB富集、提高PN反应器效能提供理论基础.
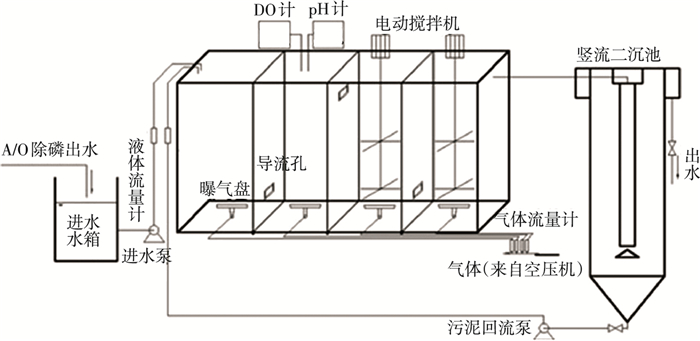
1 实验 1.1 反应器装置及进水水质反应器装置为不锈钢制的推流式反应器,后面接由有机玻璃制成的竖流式形式二沉池(图 1).主体反应器规格为2.0 m×0.6 m×1.0 m,总容积为1.2 m3,分隔成4个相同大小的格室.通过设置导流孔防止流水返混,保持反应器连续流运行所需的推流条件.A/O除磷工艺的出水用作推流式反应器进水,进行亚硝化实验.根据运行需要控制每个格室的曝气量,通过单独的气体流量计检测曝气强度.二沉池总容积为300 L,其中部分污泥进行回流,补充反应器污泥浓度.
|
图 1 亚硝化反应器装置 |
推流式亚硝化反应器接种600 L某污水厂曝气池末端的污泥(6 650 mg/L),硝化性能良好.实验用水为实际生活污水,即北方某生活小区化粪池污水经过厌氧-好氧(A/O)除磷工艺去除大部分磷、COD、BOD,反应器进水水质如表 1所示.按中国国家环保局颁布的标准方法测定进出水三氮质量浓度[4]:氨氮,纳氏试剂光度法;亚硝酸盐氮,N-(1-萘基)-乙二胺光度法;硝酸盐氮,紫外分光光度法.温度、pH及溶解氧采用WTW在线pH/DO检测.
| 表 1 实验原水水质 |
收集接种污泥及反应器启动成功后的硝化污泥,进行扫描电镜观察微观结构.方法如下:样品加入磷酸盐缓冲液(PBS,pH=8.0)洗涤3次;加入体积分数为2.5%的戊二醛,于冰箱中4 ℃条件下固定2 h;加入磷酸缓冲溶液(PBS,0.1 mol/L,pH=8.0)清洗10 min;之后进行梯度酒精脱水,体积分数分别为30%、50%、70%、90%,再用无水乙醇脱水3次,每次10 min;之后进行置换反应,即加入无水乙醇与乙酸异戊酯比为1:1及纯乙酸异戊酯各1次,每次15 min;对样品进行冷冻、真空干燥24 h、喷金,通过扫描电镜(HITACHIS-4300)观察污泥微观结构.
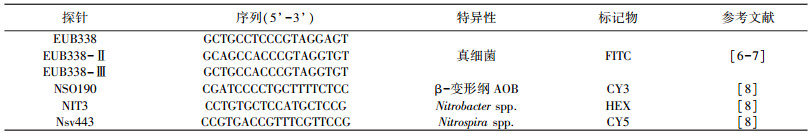
1.3 荧光原位杂交(FISH)对接种污泥与硝化污泥进行荧光原位杂交分析,所用探针如表 2所示,污泥样品经过预处理、杂交、洗涤后进行观察[5].杂交过程所用探针杂交液组成为:体积分数20%~55%的甲酰胺,0.9 mol/L NaCl,20 mmol/L Tris-HCl(pH=7.2),质量分数0.01%的SDS;杂交后洗脱液组分为:56~225 mmol/L NaCl,20 mmol/L Tris-HCl (pH=7.2),10 mmol/L EDTA,0.01%SDS.杂交后的玻片晾干后,通过荧光显微镜(BX51)及CellSens Dimension软件观察,分析杂交结果.随机捕捉20张FISH图像,利用Image-Pro Plus 6.0软件分析AOB与NOB所占比例.
| 表 2 荧光原位杂交所用16S rRNA标记的探针 |
采用改进的氯仿-SDS方法提取硝化污泥微生物基因组DNA.取1 g污泥,加入DNA提取液2.7 mL(100 mmol/L Tris-HCl,100 mmol/L EDTA,1.5 mol/L NaCl,100 mmol/L Na3PO3,质量分数为1%的CTAB,pH=8.0)、蛋白酶K 50 μL(30 mg/mL)、溶菌酶50 μL(20 mg/mL),混匀之后在37 ℃水浴、225 r/min条件下震荡30 min;加入0.3 mL质量分数为20%的SDS,65 ℃水浴2 h时,期间每隔15 min将样品管轻摇混匀.水浴完成后将离心管取出,8 000 r/min条件下离心10 min,弃沉淀;上清液加入等体积的氯仿/异戊醇(24:1),混匀后离心10 min(8 000 r/min);上清液加入0.6倍体积的异丙醇,-20 ℃冷冻过夜,再在4 ℃、10 000 r/min条件下离心5 min,去上清液,沉淀至通风处彻底晾干.加入100 μL TE(100 mmol/L Tris-HCl, 100 mmol/L EDTA,pH=8.0)溶解沉淀,-20 ℃保存备用[6].
采用通用引物GC-338F和907R扩增细菌16S rRNA基因[9].PCR反应管内加入提取的基因组DNA1.0 μL,dNTPs(2.5 mmol/L) 2.0 μL,引物GC-338F和907R(20 μmol/L)各0.5 μL,10×Buffer 2.5 μL,Ex Taq酶(5 U/μL) 0.125 μL,加入无菌水至终体积25 μL.采用TaKaRa TP600梯度PCR仪(日本)进行PCR反应,94 ℃开始变性5 min;进入循环反应,包括94 ℃、40 s,55 ℃、40 s,72 ℃、1 min 3个步骤,共运行30个循环;72 ℃终延伸10 min,4 ℃终止PCR反应.利用DNA纯化回收试剂盒(天根,中国)对PCR产物纯化回收,并通过琼脂糖凝胶电泳验证.
1.4.2 DGGE及其结果分析DGGE电泳仪器为美国Bio-Rad公司的D-Code System系统.通过加入尿素与甲酰胺配置质量分数30%~60%的变性梯度凝胶,配置质量分数为8%的聚丙烯酰胺凝胶进行PCR产物分析.DGGE操作条件为:电压120 V,电泳温度与时间分别为60 ℃与8 h,电泳缓冲液为1×TAE.电泳结束后,将凝胶清洗之后进行银染[10],在凝胶成像仪(BioRad)中通过软件Gel Doc XR获取银染后图像.
1.4.3 微生物系统发育分析通过银染,DGGE凝胶出现明显条带,对这些条带进行切割,将其溶于100 μL 1×TE(pH8.0)中,于4 ℃冰箱中过夜.以此为模板,相应不带GC夹的338F与907R为引物,扩增细菌16S rRNA基因片段.PCR片段进行纯化回收,通过T4DNA连接酶连接到载体pMD19-T (TaKaRa)上,42 ℃热击,转化到感受态细胞Escherichia coli DH5α(天根,中国)中.通过涂布平板,37 ℃过夜培养,构建目的基因克隆文库.每个DGGE条带的样品选取3~5个阳性克隆送交上海生工生物公司进行测序.获得的序列通过BLAST工具搜索相近序列,进行比对.将序列提交到GenBank,获得基因登录号为KF194203-KF194209.通过MEGA 5.0软件,以bootstrap-NJ法构建相应序列的系统发育树.
2 结果与讨论 2.1 亚硝化反应器运行效果亚硝化反应器运行效果可用氨氧化率及亚硝化率来表征,其中氨氧化率(RA)计算方式为
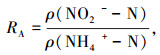
|
亚硝化率(RN)计算方式为
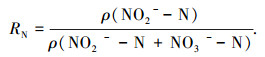
|
单位为mg/L.以亚硝化率连续7个周期超过90%作为亚硝化启动成功标志[11].
在之前的研究中已完成亚硝化反应器启动[12],过程如下:在A/O除磷反应器的出水中人工添加(NH4)2SO4至氨氮质量浓度为300 mg/L(游离氨FA为11.36 mg/L),温度为室内自然温度(16.4~25.5 ℃).采用间歇操作方式,即经过进水(0.5 h)、曝气与沉淀(1 h)、排水(1 h)、闲置5个阶段,对接种污泥进行驯化.对曝气强度采用实时控制策略[13],保持DO质量浓度0.2~0.8 mg/L,避免延时曝气.在这种工况下共运行了60个周期,出水NO2--N与NH4+-N质量比达1.0~1.30,亚硝化率一直保持90%以上,表明成功启动亚硝化反应器.之后将进水氨氮质量浓度降到80 mg/L,按SBR操作方式共运行40个周期,期间氨氧化率在90%左右,而亚硝化率一直在95%以上,说明反应器在低氨氮、间歇操作方式下,能保持亚硝化稳定运行[14].
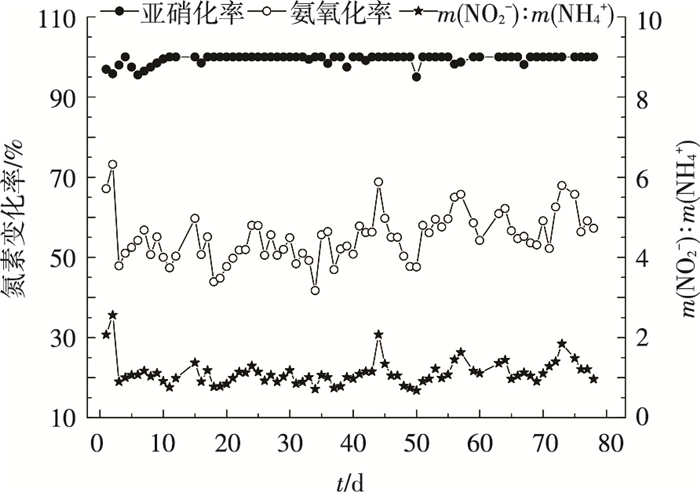
此后,反应器转变为常温低氨氮连续流运行(本文实验),进水流量(Qin)为125~160 L/h,污泥回流量(Q回)为60~80 L/h,水力停留时间(HRT)为7~9 h.调节反应器4个格室的曝气强度,分别为4~8 L/min、3~4 L/min、0(只进行缺氧搅拌)、3~5 L/min,保持反应器溶解氧(DO)质量浓度处于较低水平(0.1~0.6 mg/L).整个80 d运行期间,反应器氨氧化率、亚硝化率和出水NO2--N与NH4+-N质量比如图 2所示.
|
图 2 亚硝化反应器氨氧化率、亚硝化率与出水亚氮与氨氮比 |
由图 2可知,平均氨氮氧化率稳定在55%,亚硝化率接近100%,出水NO2--N与NH4+-N质量比稳定在1.11附近,可为后续ANAMMOX反应提供稳定水质.PN反应器亚硝化效果良好,推测AOB得到有效富集,NOB活性受到抑制.以此常温低氨氮亚硝化反应器为对象,分析其功能菌群特性.
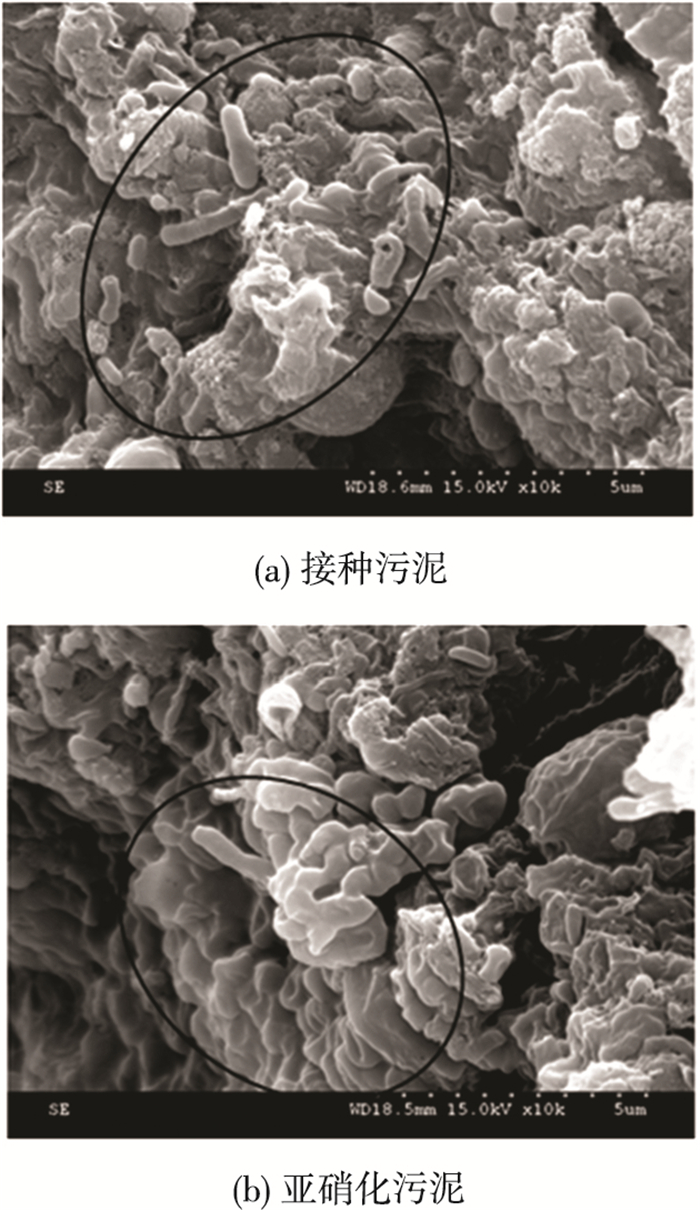
2.2 亚硝化污泥微观结构已有研究发现,Nitrosomonas类AOB数量较多时能使污泥颜色变黄,这主要是此菌体富含细胞色素所致[15].亚硝化反应器在启动过程中,就出现污泥颜色变化,由接种时浅黑色逐渐转变成浅黄色,可推测Nitrosomonas类AOB数量逐渐增多.通过扫描电镜,对接种污泥与亚硝化污泥进行微观结构对比分析,结果如图 3所示.
|
图 3 接种污泥与亚硝化污泥扫描电镜结果 |
亚硝化单胞菌属(Nitrosomonas)和亚硝化球菌属(Nitrosococcus)是在污水生物脱氮处理过程中经常出现的AOB菌属,形态分别为短杆状或球状,而NOB主要为硝化杆菌属(Nitrobacter)与硝化螺菌属(Nitrospira),对应形态分别为杆状或螺旋状.由图 3(a)可知,接种污泥中微生物种类较多,有杆状菌、短杆菌和球形菌(黑色线圈标注);而在亚硝化污泥中(图 3(b)),短杆菌、球形细菌较多(黑色线圈标注).郭建华等[16]曾经分析了短程硝化过程中种群结构的演变,SEM结果发现,接种污泥中含有杆菌、短杆菌、球形菌以及丝状菌等多种形态微生物,而随着短程硝化的启动与稳定,污泥中短杆菌和球菌数量增多,成为优势菌群,本实验亦有类似变化趋势.为进一步了解反应器内AOB与NOB丰度,开展污泥的荧光原位杂交(FISH)实验,分析AOB与NOB的相对比例关系.
2.3 反应器功能菌群FISH分析PN反应器启动成功后,亚硝化率接近100%,可推测AOB为优势菌群,NOB得到有效抑制.SEM结果也表明污泥微生物菌群结构发生了变化,但这都不能直接说明污泥中AOB或NOB数量的多寡.因此,对接种污泥与亚硝化污泥微生物进行FISH检测,对其中AOB与NOB分布及相对比例进行定量考察.选取代表性AOB、NOB荧光照片及真细菌荧光照片,通过Image-Pro Plus 6.0软件进行分析,结果如图 4所示.图 4(a)和(c)中亮点部分代表AOB(Nitrosomonas)的分布,暗色部分为真细菌.同理,图 4(b)和(d)中亮度高的部分代表NOB(Nitrobacter)的分布,颜色较暗部分为真细菌.

|
(a)和(c)中亮点部分表示Cy3标记NSO190探针与AOB(Nitrosomonas)的杂交结果,(b)和(d)中亮度较高部分表示HEX标记的NIT3探针与NOB(Nitrobacter)的杂交结果;(a)~(d)中暗色部分代表FITC标记的Eub338mix探针(EUB338、EUB338-Ⅱ和EUB338-Ⅲ)与真细菌杂交结果;标尺为20 μm. 图 4 接种污泥及亚硝化污泥荧光原位杂交分析 |
对FISH结果统计发现,接种污泥中Nitrosomonas-AOB与Nitrobacter-NOB的相对比例分别为35%和21%,说明AOB与NOB均是其中优势菌属,原因是接种污泥是从全程硝化反硝化脱氮污水处理厂中取样.本实验中,亚硝化污泥中Nitrosomonas-AOB相对比例略有升高,为37.3%,而Nitrobacter-NOB相对比例下降明显,仅为4.4%,这表明反应器中AOB成为优势菌群,而NOB逐渐淘汰.之前有研究发现,实时控制的SBR小型反应器短程硝化运行135 d后,AOB数量占绝对优势,NOB得到抑制与淘洗[16].Sinha等[17]利用FISH方法,分析了连续流系统中短程硝化前后的污泥菌群比例,发现随着反应器短程硝化的运行,AOB所占比例从接种污泥的2%~3%上升至短程硝化污泥的48%~53%,NOB比例较低,为6%~8%.这些研究表明,反应器在不同运行方式及不同控制参数影响下,AOB及NOB种群结构会发生明显变化.本实验中反应器在连续流运行时间内,AOB为优势菌群,NOB活性得到有效抑制,数量较少.
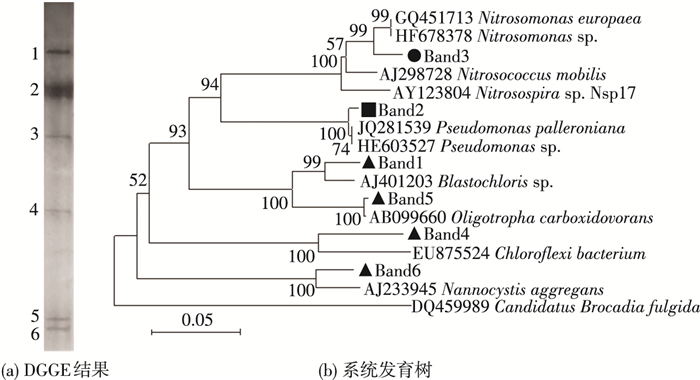
2.4 微生物群落结构解析以亚硝化污泥为对象,通过提取基因组DNA、GC338F/907R引物扩增16S rDNA基因、DGGE等步骤,分析微生物群落组成;对DGGE条带切胶、PCR、克隆测序,获得的序列进行系统发育分析,结果如图 5所示.
|
图 5 PN反应器全细菌DGGE结果与系统发育树 |
由图 5(a)可知,主要有6类优势菌群存在PN反应器内,其中条带3测序结果与Nitrosomonas sp.(HF678378)相似度为96%,表明此类氨氧化菌为PN反应器内功能菌,负责维持亚硝化过程稳定运行.条带1测序结果与Blastochloris sp.(AJ401203)相似度为96%.Blastochloris sp.为芽生绿菌属细菌,革兰氏染色为阴性,形态有杆形或卵圆形,异养厌氧生长或者微好氧生长.条带2与Pseudomonas sp.(HE603527)同源性最高,相似度为99%.Pseudomonas sp.为假单胞菌属,为常见的反硝化细菌,此类细菌的存在能够消耗一部分有机物,这可能与进水中含有少量COD相适应.
而条带4与绿弯菌门细菌Chloroflexi bacterium(EU875524)相似度为90%,该类细菌为常见的丝状菌,常在生物脱氮除磷系统中出现[18],可在活性污泥形成中起到骨架作用,有利于菌胶团的形成.条带5与α变形纲的食羧寡养菌Oligotropha carboxidovorans(AB099660)同源性最高,相似度为99%,该菌种也是活性污泥中常见的微生物[19].而条带6与δ变形纲的侏囊菌属细菌Nannocystis aggregans(AJ233945)相似度为95%,后者属于粘细菌的一种[20],该类菌为革兰氏阴性菌,属于好氧化能有机营养型细菌,难以获得纯培养.
综上,PN反应器主要以自养型Nitrosomonas sp.功能微生物为主,负责氨氧化过程;此外还存在一些异养菌,如Pseudomonas sp.、Chloroflexi bacterium及Blastochloris sp..这些异养菌的存在并没有影响反应器亚硝化效果,推测原因是进水中有机物很少(COD小于50 mg/L,BOD5小于15 mg/L),异养菌不能大量繁殖.
3 结论1) 通过高氨氮进水与低溶解氧联合抑制NOB活性方式,成功启动亚硝化反应器.此后在低基质连续流运行80 d内,继续保持低溶解氧浓度,可使反应器出水NO2--N与NH4+-N质量比稳定,获得良好的亚硝化效果.
2) PN反应器中,亚硝化污泥中以球形菌与短杆菌为主,Nitrosomonas类AOB为优势菌群,相对比例为37.3%,NOB活性被抑制,数量逐渐减少,相对比例为4.4%.
3) 对微生物群落结构进行解析,发现PN反应器存在6类优势微生物,即Blastochloris sp.、Pseudomonas sp.、Nitrosomonas sp.、Chloroflexi bacterium、Oligotropha carboxidovorans与Nannocystisaggregans,其中Nitrosomonas sp.为功能微生物AOB,可与其他微生物和谐共存,维持亚硝化反应器稳定运行.
| [1] |
KARTAL B, KUENEN J G, VAN LOOSDRECHT M C. Engineering sewage treatment with anammox[J]. Science, 2010, 328(5979): 702-703. DOI:10.1126/science.1185941 |
| [2] |
LACKNER S, GILBERT E M, VLAEMINCKS E, et al. Full-scale partial nitritation/anammox experiences-an application survey[J]. Water Research, 2014, 55: 292-303. DOI:10.1016/j.watres.2014.02.032 |
| [3] |
曾薇, 张悦, 李磊, 等. 生活污水常温处理系统中AOB与NOB竞争优势的调控[J]. 环境科学, 2009, 30(5): 1430-1436. |
| [4] |
国家环境保护总局. 水和废水监测分析方法[M]. 4版. 北京: 中国环境科学出版社, 2002.
|
| [5] |
ZHANG Z, CHEN S, WU P, et al. Start-up of the CANON process from activated sludge under salt stress in a sequencing batch biofilm reactor (SBBR)[J]. Bioresource Technology, 2010, 101(16): 6309-6314. DOI:10.1016/j.biortech.2010.03.040 |
| [6] |
曾涛涛. 常温低基质PN-ANAMMOX耦合工艺脱氮效能及微生物特性研究[D]. 哈尔滨: 哈尔滨工业大学, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10213-1013045285.htm
|
| [7] |
DAIMS H, BRÜHL A, AMANN R, et al. The domain-specific probe Eub338 is insufficient for the detection of all bacteria:development and evaluation of a more comprehensive probe set[J]. Systematic and Applied Microbiology, 1999, 22(3): 434-444. DOI:10.1016/S0723-2020(99)80053-8 |
| [8] |
MOBARRY B K, WAGNER M, URBAIN V, et al. Phylogenetic probes for analyzing abundance and spatial organization of nitrifying bacteria[J]. Applied and Environmental Microbiology, 1996, 62(6): 2156-2162. |
| [9] |
GONG Z, LIU S, YANG F, et al. Characterization of fu nctional microbial community in a membrane-aerated biofilm reactor operated for completely autotrophic nitrogen removal[J]. Bioresource Technology, 2008, 99(8): 2749-2756. DOI:10.1016/j.biortech.2007.06.040 |
| [10] |
BASSAM B J, GRESSHOFF P M. Silver staining DNA in polyacrylamide gels[J]. Nature Protocols, 2007, 2(11): 2649-54. DOI:10.1038/nprot.2007.330 |
| [11] |
李冬, 刘丽倩, 吴迪, 等. 常温低氨氮SBR亚硝化启动策略研究[J]. 中国环境科学, 2013, 33(2): 215-220. |
| [12] |
张昭, 李冬, 曾辉平, 等. 常温下部分亚硝化的启动中试研究[J]. 中国给水排水, 2012, 28(17): 21-25. DOI:10.3969/j.issn.1000-4602.2012.17.006 |
| [13] |
王淑莹, 顾升波, 杨庆, 等. SBR工艺实时控制策略研究进展[J]. 环境科学学报, 2009, 29(6): 1121-1130. |
| [14] |
张昭, 李冬, 邱文新, 等. 城市污水部分亚硝化的实现与稳定运行[J]. 中南大学学报(自然科学版), 2013, 44(7): 3066-3071. |
| [15] |
郑平, 徐向阳, 胡宝兰. 新型生物脱氮理论与技术[M]. 北京: 科学出版社, 2004.
|
| [16] |
郭建华, 王淑莹, 郑雅楠, 等. 实时控制实现短程硝化过程中种群结构的演变[J]. 哈尔滨工业大学学报, 2010, 42(8): 1259-1263. DOI:10.11918/j.issn.0367-6234.2010.08.017 |
| [17] |
SINHA B, ANNACHHATRE A P. Assessment of partial nitrification reactor performance through microbial population shift using quinone profile, FISH and SEM[J]. Bioresource Technology, 2007, 98(18): 3602-3610. DOI:10.1016/j.biortech.2006.11.034 |
| [18] |
YOON D N, PARK S J, KIM S J, et al. Isolation, characterization, and abundance of filamentous members of caldilineae in activated sludge[J]. Journal of Microbiology, 2010, 48(3): 275-283. DOI:10.1007/s12275-010-9366-8 |
| [19] |
MALIK A, SAKAMOTO M, HANAZAKI S, et al. Coaggregation among nonflocculating bacteria isolated from activated sludge[J]. Applied and Environmental Microbiology, 2003, 69(10): 6056-6063. DOI:10.1128/AEM.69.10.6056-6063.2003 |
| [20] |
SPROER C, REICHENBACH H, STACKEBRANDT E. The correlation between morphological and phylogenetic classification of myxobacteria[J]. International Journal of Systematic Bacteriology, 1999, 49(3): 1255-1262. DOI:10.1099/00207713-49-3-1255 |
 2016, Vol. 48
2016, Vol. 48