2. 北京工业大学国家工程实验室,北京市水质科学与水环境恢复工程重点实验室,北京 100124
2. National Engineering Laboratory for Advanced Municipal Wastewater Treatment and Reuse Technology, Key Laboratory of Beijing for Water Quality Science and Water Environment Recovery Engineering (Beijing University of Technology), Beijing 100124, China
在污水处理中,由于受到进水水质、水量波动的影响,生长在其中的细菌不可避免地会经历交替的底物丰富期和匮乏期.当底物匮乏时,不同细菌会表现出不同的饥饿应对机制[1],而不能够适应饥饿环境的细菌往往会被淘汰.目前,已有许多研究学者采用分子生物学手段考察了污水处理系统中微生物在饥饿条件下的种群结构变化.例如,Elawwad等[2]采用末端限制性片段长度多态性技术发现生物膜反应器中的优势氨氧化菌(ammonium oxidizing bacteria, AOB)在饥饿过程中会逐步转变为Nitrosomonas europaea/eutropha;胡怡杉等[3]采用变性梯度凝胶电泳技术发现在饥饿条件下膜生物反应器和传统活性污泥反应器中的微生物均会发生明显的种群结构变化.但这些报道所用的传统检测手段由于低通量限制可能会造成微生物多样性检测的偏差[4].高通量测序技术具有测序通量高、周期短等优点,已逐渐用于检测活性污泥系统中微生物的多样性[4-5].然而,利用高通量测序技术对活性污泥在好氧饥饿条件下的微生物种群结构变化的研究鲜有报道.
本研究将普通序批式反应器(sequencing batch reactor, SBR)处理生活污水的活性污泥置于好氧饥饿环境中,利用Illumina高通量测序和实时定量PCR(real-time quantitative PCR, qPCR)技术研究了活性污泥在好氧饥饿过程中细菌群落及氮磷相关功能菌的丰度变化,并考察了活性污泥在好氧饥饿后硝化及除磷性能的恢复情况.
1 实验 1.1 活性污泥来源及特性有效容积为10 L的SBR反应器作为母反应器(SBRPR),其运行方式为进水10 min、厌氧搅拌30 min、好氧曝气240 min、外加甲醇(体积分数5% 10 mL)进行缺氧搅拌90 min、好氧曝气45 min、沉淀55 min和出水10 min,1 d运行3个周期,1个周期为8 h.进水为实际生活污水(具体水质指标见表 1),通过一个周期运行,可去除99%以上的氮磷等污染物质.该工艺水力停留时间(hydraulic retention time, HRT)和污泥停留时间(sludge retention time, SRT)分别为13.8 h和30 d.
| 表 1 实验用水水质 Tab. 1 Characteristics of the trial wastewater |
好氧饥饿试验采用4个有效容积为3 L的SBR反应器,在母反应器好氧阶段结束后取泥,采用SBRPR出水清洗和稀释使得各好氧饥饿批次反应器的接种污泥质量浓度均为5 g/L左右.好氧饥饿时间分别为3,7,14和30 d,所对应的好氧饥饿批次反应器分别为SBR1、SBR2、SBR3和SBR4.在好氧饥饿阶段,各反应器进行持续搅拌和曝气,DO质量浓度维持在(8.50±1.0)mg/L;温度保持室温,平均温度维持在(20±2)℃;pH不加控制,在好氧饥饿10 d后下降至5.30,18 d后上升至6.88,30 d后下降至5.00维持稳定.
好氧饥饿结束后,采用SBRPR出水离心清洗去除各批次反应器中活性污泥在好氧饥饿条件下释放的氮和磷物质.由于好氧饥饿对于活性污泥的减量效果,清洗稀释后各反应器的污泥质量浓度分别为4 304,3 708,2 628和2 176 mg/L,且污泥中有机质含量逐步减少,VSS与TSS比由原来的0.86下降到0.78.恢复期以实际生活污水作为各批次反应器的进水,运行方式为进水20 min (1.5 L)、好氧曝气120 min、外加碳源(体积分数5%甲醇1.3 mL)缺氧搅拌60 min、沉淀90 min、出水20 min和闲置50 min,1 d运行4个周期,1个周期为6 h.采用空气压缩机进行曝气,气体流量均控制在600 mL/min.在23 d的恢复过程中,各批次反应器每天在缺氧阶段结束后手动排泥100 mL,同时,考虑到出水及取样过程中损失的污泥量,计算可得各批次反应器的SRT均为15 d左右,同时各批次反应器的HRT均为11.67 h.
1.3 DNA的提取为测量好氧饥饿前后活性污泥的微生物种群结构变化,在好氧饥饿试验开始时和结束后分别取活性污泥样品.样品通过冷冻干燥处理后保存,利用DNA提取试剂盒(Fast DNA Spin kit for soil, BIO 101 system, USA)进行总DNA的提取,提取方法见试剂盒说明,提取完毕后利用ND-1000紫外分光光度计(NanoDrop Technologies, Wilmington, DE, USA)检测DNA提取效果及浓度.
1.4 高通量测序和分析采用Illumina Miseq PE300(上海美吉生物科技)高通量测序平台进行活性污泥好氧饥饿前后的微生物菌群结构演化研究,针对细菌16S rRNA V4-V5区对各活性污泥样品进行PCR扩增,所用引物为338F(ACTCCTACGGGAGGCAGCA)和806R(GGAC-TACHVGGGTWTCTAAT). PCR扩增的20 μL体系配置如下:4 μL 5×FastPfu Buffer,2 μL dNTPs,0.4 μL前/后引物,0.4 μL FastPfu Primer,10 ng样品DNA,并用ddH2O补齐20 μL.反应条件为:预加热95 ℃,2 min;随后进行25周期扩增反应(变性95 ℃,30 s;退火55 ℃,30 s;延伸72 ℃,40 s);终止延伸72 ℃,10 min.获得扩增产物后,利用AxyPrep DNA Gel Extraction Kit试剂盒(Axygen Biosciences美国)进行切胶纯化,并将其溶于30 μL DNase-free H2O.进一步通过2.0%琼脂糖凝胶电泳检测PCR产物纯化效果,测定纯化后PCR产物的浓度.之后,将DNA扩增纯化样品利用Illumina MiSeq测序仪进行上机测序.
原始fastq文件采用QIIME 1.17版工具包进行序列拼接和过滤,剔除低质量序列和无法比对的序列,最后各样本所得到的高通量序列数分别为54599(Day 0),57248(Day 3),50171(Day 7),46669(Day 14)和43385(Day 30).所测得的原始序列数据已上传到NCBI中Sequence Read Archive数据库,项目编号分别为SRR5146098,SRR5146099,SRR5146100,SRR5146101和SRR5146102.为了在同一测序深度上对不同的样品进行分析,采用MOTHUR v.1.30.1程序将样品进行最小序列值归一化(38673).利用软件Usearch 7.1按照97%相似性进行统计注释和比对聚类,并通过与数据库Silva最新序列比对定义OTUs(operational taxonomic units),最后利用RDP(ribosomal database project)classifier贝叶斯算法将OTUs代表序列在各分类水平上分析种属组成及丰度关系.
1.5 实时定量PCR采用SYBR Green实时定量PCR技术检测活性污泥中AOB、亚硝酸盐氧化菌(nitrite oxidizing oacteria, NOB; 包括Nitrobacter spp.和Nitrospira spp.)和全菌的菌群绝对丰度值.扩增体系为20 μL,该体系中包括7 μL去离子水,10 μL SYBR® Premix Ex TaqTM(Takara,大连),0.4 μL ROX Reference Dye 50,0.3 μL的前引物和后引物(10 μmol/L),2 μL的DNA样品.扩增条件及各引物的寡核苷酸探针序列参照文献[6].为确保数据结果的有效性,标准曲线是将已知浓度的质粒DNA进行10倍梯度稀释建立完成,标准品和待测样均设3个平行,同时设置阴性对照,要求相关系数R2大于0.99,扩增效率在90%~110%.
1.6 其他分析测试方法水样中的NH4+-N、NO2--N、NO3--N和PO43--P采用Quik Chem® 8500流动注射全自动分析仪(美国LACHAT公司)测定;SCOD、TN、MLSS和MLVSS均采用APHA的标准方法[7]测定;pH、DO和温度采用德国WTW Multi 340i便携式检测仪及其探头检测.
2 结果与讨论 2.1 好氧饥饿对活性污泥硝化及除磷性能的影响图 1为不同好氧饥饿时间后各批次反应器内活性污泥的硝化和除磷性能的恢复情况,包括了各反应器内的NH4+-N去除率、PO43--P去除率和好氧末的亚硝酸盐积累率(RNA, RNA=ρ(NO2--N)/(ρ(NO3--N)+ρ(NO2--N))×100%).好氧饥饿前,SBRPR的活性污泥在长达6个月的运行过程中,取得了良好的污泥性状,系统出水的NH4+-N、NO2--N、NO3--N和PO43--P均低于0.1 mg/L,不过系统在好氧阶段的硝化产物主要以NO3-为主,好氧末计算所得的RNA仅为0.2%.好氧饥饿后,活性污泥的硝化和除磷性能均产生了一定的下降,且活性下降程度与好氧饥饿时间成正比.由图 1可知,在恢复期第1天,SBR1、SBR2、SBR3和SBR4的NH4+-N去除率分别为38.9%,14.3%,8.7%和1.0%,PO43--P去除率分别为68.9%,54.6%,11.6%和8.4%.此外,好氧饥饿时间越短,污泥的活性恢复越快.由图 1(a)可知,SBR1反应器污泥硝化和除磷性能的恢复最快,分别在第3天和第4天恢复到最高水平,平均NH4+-N去除率和平均PO43--P去除率分别达(73.2±2.0)%和(99.3±1.2)%.SBR2反应器的污泥硝化和除磷性能的恢复次之,其PO43--P去除率在第7天恢复到最高水平(99.2%),但污泥硝化性能的恢复速度相对较慢,平均NH4+-N去除率在恢复期末达(61.4±3.0)%.此外,SBR3和SBR4反应器在长达23 d的恢复过程中,其NH4+-N去除率和PO43--P去除率均未达到SBR1反应器的水平.这说明活性污泥处于好氧饥饿环境中的时间过长,会对污泥的硝化和除磷性能造成较大的不利影响,进而影响系统的脱氮除磷效果.
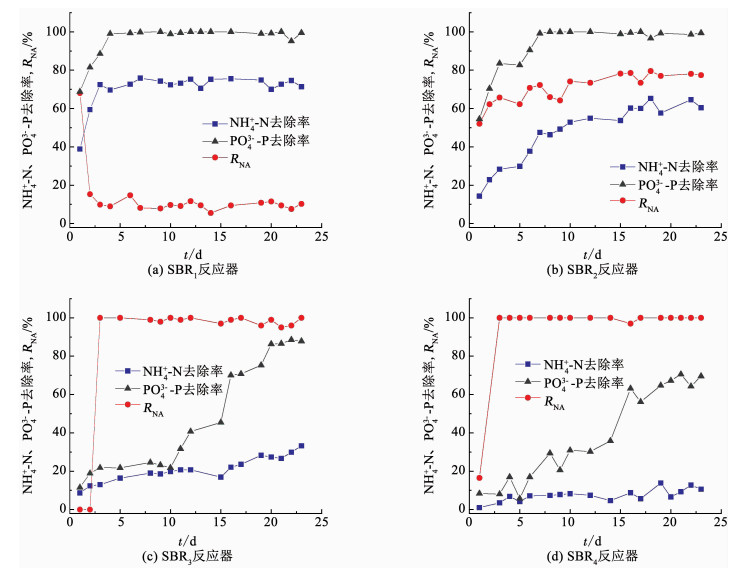
|
图 1 恢复期间活性污泥的NH4+-N、PO43--P去除率和RNA变化 Fig. 1 Variations of NH4+-N, PO43--P removal efficiency, and RNA during resuscitation period after different aerobic starvation times |
好氧饥饿时间越长,系统在恢复阶段的亚硝积累效果越好.由图 1可知,14 d和30 d的好氧饥饿处理后,SBR3和SBR4反应器在恢复期的RNA基本维持在99.0%左右,好氧末的硝化产物均以NO2-为主.然而,由于较短的好氧饥饿时间(3 d),SBR1反应器系统中的NOB没有得到充分抑制,当环境中存在NO2-时,NOB能够利用NO2-进行快速恢复,进而导致SBR1反应器的RNA由启动初期的68.0%快速下降到9.3%.相比SBR1,SBR2反应器中的亚硝积累效果较为稳定,系统在恢复阶段取得了RNA稳定在70%以上的短程硝化,同时,系统的硝化和除磷性能均得到较为明显的恢复.这说明7 d的好氧饥饿时间可有效抑制系统中NOB的活性,同时维持了较好的污泥硝化和除磷性能,确保了系统的再次启动.过长的好氧饥饿时间虽然能获得较高的亚硝酸盐积累效果,但容易破坏系统的硝化和除磷性能,延长系统的恢复时间;而过短的好氧饥饿时间又不能有效地抑制NOB的活性,无法实现系统的短程硝化.
2.2 微生物群落丰度与多样性分析采用高通量测序检测了活性污泥在好氧饥饿前后的微生物菌群结构变化.污泥样本所获得的有效序列通过与Silva数据库比对划分,共获得1 359个OTUs,其中各样本的OTUs数分别为1 012(Day 0),1 084(Day 3),1 090(Day 7),884(Day 14)和771(Day 30).微生物种群分布中Alpha多样性相关的各项指标如表 2所示.5个污泥样本的Coverage值均大于99%,说明绝大部分的污泥样品基因序列在测序后被组装得到,测序结果能够较准确地反映样品的微生物特性.此外,各样本的稀缺性曲线也都趋于平坦(图 2(a)),说明测序深度可以覆盖样品中绝大多数的微生物信息,测序数据量合理.
| 表 2 污泥样本的Alpha多样性指数(97%相似水平) Tab. 2 Alpha diversity indices for the five samples with a threshold of 0.97 |
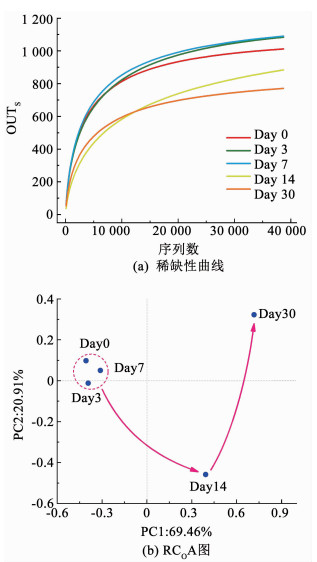
|
图 2 细菌稀缺性曲线与PCoA图 Fig. 2 Rarefaction curves and PCoA diagrams of the bacterial communities |
细菌被淘洗,菌群的多样性大幅度下降.好氧饥饿30 d后,Shannon指数逐步由3.32上升到4.52,Simpson指数逐步由0.208 3下降到0.041 3,说明伴随着好氧饥饿的进行,适应环境的某些细菌大量增殖,菌群多样性和均匀性得到改善. 30 d的好氧饥饿环境中,微生物种群中的优势菌群经历了一个动态变化过程,既有原有的优势种群逐步消亡,又有适应好氧饥饿环境的菌种增强成为新的优势菌群.
图 2(b)采用主坐标分析(PCoA)研究5个污泥样本群落组成之间的相似性或差异性[10].可以看出,好氧饥饿前7 d的3个污泥样本较为接近,相似性较强,这同样说明短期(7 d)的好氧饥饿环境并不会显著地改变活性污泥的种群结构;好氧饥饿14和30 d的污泥样本与前7 d的3个污泥样本相距较远,说明好氧饥饿超过两周以后,污泥的种群结构发生了明显变化.基于OTUs分类的Venn图分析(图 3)也同样指出,随着好氧饥饿的进行,Day14和Day30样本所共有的OTUs占总OTUs的比例分别为69.39%,68.58%,47.68%和35.25%;从各污泥样本独有的OTUs来看,好氧饥活性污泥的相似性逐渐减少,差异性逐渐增强. 30 d的好氧饥饿过程中,5个污泥样本共有的OTUs仅占总数的28.99%,Day0样本与Day3、Day7、饿30 d后的污泥样本所独有的OTUs为62个,远高于其他样本Day0(6)、Day3(7)、Day7(4)和Day14(13).说明长期好氧饥饿环境促进了某些原本并不存在的微生物生长,使得污泥的种群结构发生了极大的改变.

|
图 3 污泥样本基于OTUs分类的Venn图 Fig. 3 Venn of bacterial communities from the five samples based on the classified OTUs |
图 4分别从门、纲和属的水平分析了活性污泥在30 d好氧饥饿过程中的微生物种群结构变化.5个活性污泥样本中相对丰度大于1%的菌门共检测到15个,其中变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和厚壁菌门(Firmicutes)为各样本的主要优势菌门,这三者的相对丰度之和在5个污泥样本中的比例分别为73.52%,77.81%,72.11%,88.27%和85.05%.由图 4(a)可知,Proteobacteria作为以城市污水为原水的活性污泥中最主要的菌门[11],其在好氧饥饿前7 d的相对丰度基本稳定在52.00%左右,14 d后快速上升到73.86%,30 d后又迅速下降到36.78%.这说明Proteobacteria对于好氧饥饿环境有着较强的抵抗能力,能够抵抗短期的好氧饥饿冲击.从纲水平可以看出,Proteobacteria主要由α-Proteobacteria、β-Proteobacteria、γ-Proteobacteria、δ-Proteobacteria和ε-Proteobacteria 5类组成,其在30 d好氧饥饿过程中的相对丰度变化主要由β-Proteobacteria和δ-Proteobacteria这两类菌的丰度变化所贡献(图 4(b)). Bacteroidetes作为活性污泥中的第二大类菌门,在5个污泥样本中的比例分别为22.35%,23.07%,18.66%,11.44%和18.96%,其相对丰度变化与Proteobacteria表现出相反的趋势.由纲水平可知,Bacteroidetes中含量最大的菌纲为鞘脂杆菌纲(Sphingobacteria)(图 4(b)).有研究表明,Sphingobacteria作为一种能够产生鞘脂的细菌,包括聚糖菌等细菌[12].正由于其特殊的鞘脂结构和体内富集的PHA等高能物质的存在,其对于好氧饥饿环境有着较强的抵抗能力,故而可以在长期好氧饥饿环境中得以生存.厚壁菌门(Firmicutes)作为一类低G+C含量的革兰氏阳性菌,有研究表明其在污泥高温好氧消化工艺中发挥着重要作用[13].好氧饥饿处理30 d后,其含量由最初的0.82%逐步上升到29.31%,进而成为活性污泥中仅次于Proteobacteria的第二大类菌门.由纲水平可见,Firmicutes主要由梭菌纲(Clostridia)、杆菌纲(Bacilli)和产芽孢菌纲(Erysipelotrichia)构成,其中Clostridia和Bacilli在好氧饥饿第30天分别上升到14.51%和12.43%(图 4(b)).此外,从属水平可以看出,好氧饥饿30 d后,芽孢杆菌属(Bacillus)、乳球菌属(Lactococcus)、巴斯德氏芽菌属(Pasteuria)、Romboutsia、梭菌属(Clostridium_sensu_stricto_1)、肠杆菌属(Intestinibacter)和曲霉菌属(Turicibacter)等属于Firmicutes的一些细菌大量增殖,逐步成为活性污泥系统中的优势菌属(图 4(c)).

|
图 4 污泥样本中含有大于1%的主要细菌序列的相对丰度(门、纲和属的水平) Fig. 4 Relative abundances of the major bacterial sequences (>1% in any sample) from the five samples at the phylum level, class level, and genus level |
常见的具有除磷能力的细菌[14-15]如聚磷菌(Candidatus_Accumlibater)、红环菌(Rhodocyclaeae group)、不动杆菌(Acinetobacter)和反硝化聚磷菌(Dechloromonas)等细菌在本污泥样本中含量很低,且含量随着好氧饥饿的进行而逐步减少.然而,系统除磷性能的恢复明显快于硝化性能(图 1),这可能是由于除磷菌作为异养菌,具有合成胞内储能物质的能力,在好氧饥饿过程中,能够利用体内储存的PHA、糖原和聚磷颗粒作为能量来源,维持一定的细胞活性[16].因此,相比自养硝化菌,除磷菌这类异养菌能够更快地从饥饿环境中恢复.陶厄氏菌属(Thauera)和侏囊菌亚目(Haliangium)属于变形菌门,有研究表明这两类菌属于反硝化细菌[17].由图 4(c)可知,经过14 d好氧饥饿后,在活性污泥中这两类菌逐步占据优势地位,相对丰度分别达44.42%和12.32%,之后由于好氧饥饿后期Firmicutes菌群的大量增殖,导致其相对丰度逐步减少.
亚硝化单胞菌属(Nitrosomonas)属于Proteobacteria在本研究中作为优势AOB菌,发挥着将进水NH4+氧化为NO2-的作用;硝化螺菌属(Nitrospira)属于硝化螺旋菌门作为优势NOB菌,发挥着将NO2-转化为NO3-的作用.此外,本研究中属于AOB菌的还有亚硝化单胞菌科(Nitrosomona- daceae_uncultured),属于NOB菌的还有Candidatus _ Nitrotoga、硝化刺菌属(Nitrospina)和硝化刺菌科(Nitrospinaceae_uncultured).与前人研究[5]一致,本研究并没与检测到常见的属于NOB菌的硝化杆菌属(Nitrobacter).30 d好氧饥饿过程中,Nitrosomonas和Nitrospira的相对丰度在好氧饥饿前期(7 d)分别上升至1.06%和5.22%,之后逐步下降到0.03%和0.86%(图 5(a)).为验证高通量数据的可靠性,本研究同时采用qPCR技术考察了好氧饥饿过程中AOB、Nitrobacter和Nitrospira的相对丰度变化.qPCR的结果同样表明,AOB、Nitrobacter和Nitrospira的相对丰度均在好氧饥饿第7天达到最大值,分别为0.78%,0.98%和2.33%(图 5(b)).相比高通量数据,qPCR所测得的较低的AOB和NOB拷贝数可能是由于传统分子生物学手段无法获得充足的菌群信息,进而无法覆盖完整的菌群结构[4, 18].
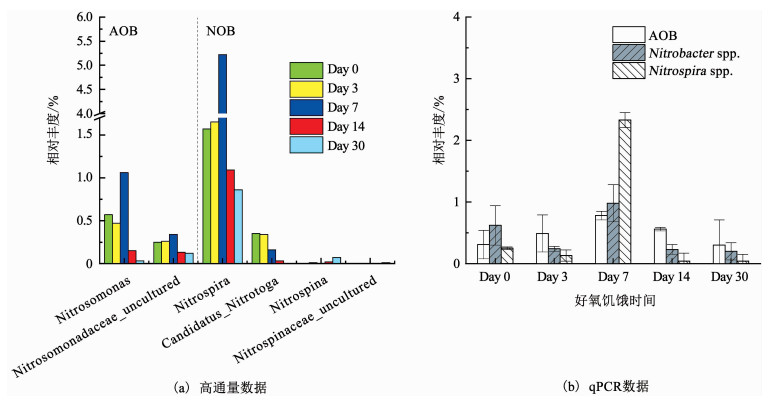
|
图 5 好氧饥饿过程中硝化细菌的相对丰度变化 Fig. 5 Variations of the relative abundances for the nitrifying bacteria during aerobic starvation period |
好氧饥饿初期,AOB和NOB相对丰度的上升可能是由于好氧饥饿过程中细菌衰亡裂解释放了大量的有机氮,后转化为NH4+为AOB提供了底物,同时,NOB能够利用AOB产生的NO2-进行细胞维持.因此,SBR1和SBR2的硝化性能能够快速恢复,其中SBR2相比于SBR1较高的RNA是由于AOB较NOB具有更快的活性恢复能力[6].好氧饥饿14 d后,AOB和NOB相对丰度的迅速下降主要是由于系统中较低的pH抑制了硝化细菌的生长[19],故而SBR3和SBR4硝化性能的恢复较为缓慢.对于污水处理工艺而言,过长的好氧饥饿环境不利于系统硝化和除磷性能的维持,在实际运行过程中应避免系统处于长期的好氧饥饿环境中,不过短期的好氧饥饿(7 d)可在保障系统硝化及除磷性能的同时,改变系统的硝化类型,促进系统由全程硝化向短程硝化转变.
3 结论1) 基于OTUs的Alpha多样性分析,污泥的种群结构在短期(7 d)好氧饥饿条件下变化不大,但随好氧饥饿时间的延长,污泥的种群结构差异变得明显.
2) 30 d好氧饥饿过程中Proteobacteria和Bacteroidetes等原有优势菌门的丰度逐步降低,而适应了好氧饥饿环境的Firmicutes逐步增殖成为优势菌门.
3) 氮磷相关功能细菌均随好氧饥饿的进行而逐步衰亡,不过硝化菌在好氧饥饿初期可利用细菌衰亡裂解释放的氨氮进行细胞维持,确保了短期好氧饥饿系统硝化性能的快速恢复.
| [1] |
HAO Xiaodi, WANG Qilin, ZHU Jingyi, et al. Microbiological endogenous processes in biological wastewater treatment systems[J]. Critical Reviews in Environmental Science and Technology, 2010, 40(3): 239. DOI:10.1080/10643380802278901 |
| [2] |
ELAWWAD A, SANDNER H, KAPPELMEYER U, et al. Long-term starvation and subsequent recovery of nitrifiers in aerated submerged fixed-bed biofilm reactors[J]. Environmental Technology, 2013, 34(8): 945. DOI:10.1080/09593330.2012.722758 |
| [3] |
胡怡杉, 孙宝盛, 王盛勇. MBR和CAS工艺污泥在贫营养培养条件下的微生物群落结构研究[J]. 环境科学学报, 2011, 31(9): 1900. HU Yishan, SUN Baosheng, WANG Shengyong. Microbial community structure of sludge in MBR and CAS under oligotrophic environment[J]. Acta Scientiae Circumstantiae, 2011, 31(9): 1900. |
| [4] |
WANG Bo, PENG Yongzhen, GUO Yuanyuan, et al. Illumina MiSeq sequencing reveals the key microorganisms involved in partial nitritation followed by simultaneous sludge fermentation, denitrification and anammox process[J]. Bioresource Technology, 2016, 207: 118. DOI:10.1016/j.biortech.2016.01.072 |
| [5] |
YE Lin, SHAO Mingfei, ZHANG Tong, et al. Analysis of the bacterial community in a laboratory-scale nitrification reactor and a wastewater treatment plant by 454-pyrosequencing[J]. Water Research, 2011, 45(15): 4390. DOI:10.1016/j.watres.2011.05.028 |
| [6] |
LIU Wenlong, YANG Qing, MA Bin, et al. Rapid achievement of nitritation using aerobic starvation[J]. Environmental Science & Technology, 2017, 51: 4001. DOI:10.1021/acs.est.6b04598 |
| [7] |
Federation Water Environmental. Standard methods for the examination of water and wastewater[M]. Washington D C: American Public Health Association (APHA), 2005.
|
| [8] |
BOLLMANN A, SCHMIDT I, SAUNDSES A M, et al. Influence of starvation on potential ammonia-oxidizing activity and amoA mRNA levels of Nitrosospira briensis[J]. Applied and Environmental Microbiology, 2005, 71(3): 1276. DOI:10.1128/AEM.71.3.1276-1282.2005 |
| [9] |
SMITH N R, YU Zhongtang, MOHN W W. Stability of the bacterial community in a pulp mill effluent treatment system during normal operation and a system shutdown[J]. Water Research, 2003, 37(20): 4873. DOI:10.1016/j.watres.2003.08.019 |
| [10] |
LIANG Bin, CHENG Haoyi, VAN NOSTRAND J D, et al. Microbial community structure and function of nitrobenzene reduction biocathode in response to carbon source switchover[J]. Water Research, 2014, 54: 137. DOI:10.1016/j.watres.2014.01.052 |
| [11] |
ZHANG Tong, SHAO Mingfei, YE Lin. 454 pyrosequencing reveals bacterial diversity of activated sludge from 14 sewage treatment plants[J]. The ISME Journal, 2012, 6: 1137. DOI:10.1038/ismej.2011.188 |
| [12] |
WEISSBRODT D G, MAILLARD J, BROVELLI A, et al. Multilevel correlations in the biological phosphorus removal process: From bacterial enrichment to conductivity-based metabolic batch tests and polyphosphatase assays[J]. Biotechnology and Bioengineering, 2014, 111(12): 2421. DOI:10.1002/bit.25320 |
| [13] |
PITERINA A V, BARTLETT J, PEMBROKE J T. Phylogenetic analysis of the bacterial community in a full scale autothermal thermophilic aerobic digester (ATAD) treating mixed domestic wastewater sludge for land spread[J]. Water Research, 2012, 46(8): 2488. DOI:10.1016/j.watres.2012.01.045 |
| [14] |
LI Dong, LV Yufeng, ZENG Huiping, et al. Long term operation of continuous-flow system with enhanced biological phosphorus removal granules at different COD loading[J]. Bioresource Technology, 2016, 216: 761. DOI:10.1016/j.biortech.2016.06.022 |
| [15] |
ZHANG Miao, YANG Qing, ZHANG Jianhua, et al. Enhancement of denitrifying phosphorus removal and microbial community of long-term operation in an anaerobic anoxic oxic-biological contact oxidation system[J]. Journal of Bioscience and Bioengineering, 2016, 122(4): 456. DOI:10.1016/j.jbiosc.2016.03.019 |
| [16] |
LU Huabing, KELLER J, YUAN Zhiguo. Endogenous metabolism of Candidatus Accumulibacter phosphatis under various starvation conditions[J]. Water Research, 2007, 41: 4646. DOI:10.1016/j.watres.2007.06.046 |
| [17] |
MCLLROY S J, STARNAWSKA A, STARNAWSKI P, et al. Identification of active denitrifiers in full-scale nutrient removal wastewater treatment systems[J]. Environment Microbiology, 2014, 18(1): 50. DOI:10.1111/1462-2920.12614 |
| [18] |
LU Lu, XING Defeng, REN Nanqi. Pyrosequencing reveals highly diverse microbial communities in microbial electrolysis cells involved in enhanced H2 production from waste activated sludge[J]. Water Research, 2012, 46(7): 2425. DOI:10.1016/j.watres.2012.02.005 |
| [19] |
WETT B, RAUCH W. The role of inorganic carbon limitation in biological nitrogen removal of extremely ammonia concentrated wastewater[J]. Water Research, 2003, 37(5): 1100. DOI:10.1016/S0043-1354(2)00440-2 |
 2019, Vol. 51
2019, Vol. 51


